Mol:FL5FAAGA0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 69 0 0 0 0 0 0 0 0999 V2000 | + | 63 69 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1179 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1179 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1179 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1179 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4031 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4031 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6884 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6884 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6884 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6884 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4031 1.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4031 1.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0264 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0264 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7412 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7412 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7412 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7412 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0264 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0264 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0264 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0264 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6412 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6412 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3697 1.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3697 1.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0981 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0981 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0981 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0981 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3697 3.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3697 3.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6412 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6412 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4031 -0.6088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4031 -0.6088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8779 3.3171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8779 3.3171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4649 0.1028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4649 0.1028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9717 1.9472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9717 1.9472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3872 2.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3872 2.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7124 1.7582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7124 1.7582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9264 2.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9264 2.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7124 3.2612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7124 3.2612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3872 3.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3872 3.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1731 2.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1731 2.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1334 4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1334 4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6818 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6818 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8612 3.3310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8612 3.3310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0178 2.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0178 2.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2631 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2631 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4424 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4424 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0322 -1.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0322 -1.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2339 -2.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2339 -2.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0548 -2.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0548 -2.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4648 -1.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4648 -1.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8715 -0.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8715 -0.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1172 -1.7418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1172 -1.7418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7295 -1.6725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7295 -1.6725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6719 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6719 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6170 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6170 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2918 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2918 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9350 0.5821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9350 0.5821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6257 0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6257 0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9875 1.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9875 1.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4482 0.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4482 0.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7350 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7350 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7026 0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7026 0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0178 1.1301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0178 1.1301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3180 -3.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3180 -3.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0080 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0080 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7873 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7873 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2272 -3.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2272 -3.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0358 -2.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0358 -2.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2565 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2565 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8165 -3.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8165 -3.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2567 -1.9781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2567 -1.9781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7463 -2.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7463 -2.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0459 -2.9353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0459 -2.9353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4271 -4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4271 -4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5459 1.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5459 1.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3143 2.7102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3143 2.7102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 43 48 1 0 0 0 0 | + | 43 48 1 0 0 0 0 |
| − | 44 49 1 0 0 0 0 | + | 44 49 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 45 50 1 0 0 0 0 | + | 45 50 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 41 51 1 0 0 0 0 | + | 41 51 1 0 0 0 0 |
| − | 53 52 1 1 0 0 0 | + | 53 52 1 1 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 52 1 1 0 0 0 | + | 57 52 1 1 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 55 59 1 0 0 0 0 | + | 55 59 1 0 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 52 61 1 0 0 0 0 | + | 52 61 1 0 0 0 0 |
| − | 53 51 1 0 0 0 0 | + | 53 51 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 46 62 1 0 0 0 0 | + | 46 62 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 69 | + | M SBL 1 1 69 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 69 -0.5584 -0.3025 | + | M SBV 1 69 -0.5584 -0.3025 |
| − | S SKP 5 | + | S SKP 5 |
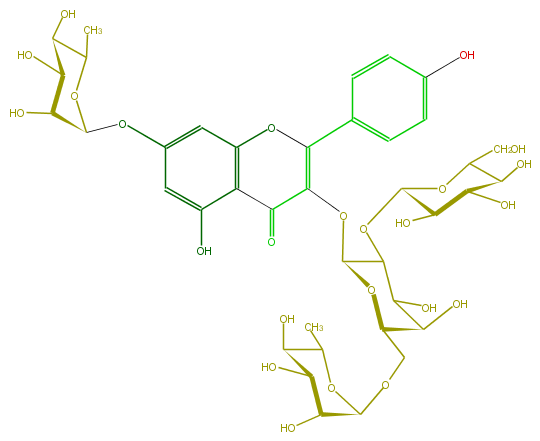
| − | ID FL5FAAGA0026 | + | ID FL5FAAGA0026 |
| − | FORMULA C39H50O24 | + | FORMULA C39H50O24 |
| − | EXACTMASS 902.269202528 | + | EXACTMASS 902.269202528 |
| − | AVERAGEMASS 902.7999 | + | AVERAGEMASS 902.7999 |
| − | SMILES O(C(O7)C(C(C(O)C(CO)7)O)O)C(C(OC(C6=O)=C(Oc(c46)cc(OC(O5)C(C(O)C(O)C5C)O)cc4O)c(c3)ccc(c3)O)1)C(C(C(COC(O2)C(C(O)C(C2C)O)O)O1)O)O | + | SMILES O(C(O7)C(C(C(O)C(CO)7)O)O)C(C(OC(C6=O)=C(Oc(c46)cc(OC(O5)C(C(O)C(O)C5C)O)cc4O)c(c3)ccc(c3)O)1)C(C(C(COC(O2)C(C(O)C(C2C)O)O)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 69 0 0 0 0 0 0 0 0999 V2000
-2.1179 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1179 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4031 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6884 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6884 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4031 1.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0264 0.2162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7412 0.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7412 1.4543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0264 1.8669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0264 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6412 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3697 1.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0981 2.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0981 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3697 3.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6412 2.8670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4031 -0.6088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8779 3.3171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4649 0.1028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9717 1.9472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3872 2.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7124 1.7582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9264 2.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7124 3.2612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3872 3.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1731 2.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1334 4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6818 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8612 3.3310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0178 2.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2631 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4424 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0322 -1.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2339 -2.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0548 -2.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4648 -1.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8715 -0.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1172 -1.7418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7295 -1.6725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6719 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6170 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2918 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9350 0.5821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6257 0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9875 1.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4482 0.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7350 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7026 0.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0178 1.1301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3180 -3.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0080 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7873 -3.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2272 -3.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0358 -2.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2565 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8165 -3.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2567 -1.9781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7463 -2.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0459 -2.9353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4271 -4.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5459 1.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3143 2.7102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
27 30 1 0 0 0 0
22 31 1 0 0 0 0
23 21 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
43 48 1 0 0 0 0
44 49 1 0 0 0 0
38 42 1 0 0 0 0
45 50 1 0 0 0 0
33 20 1 0 0 0 0
41 51 1 0 0 0 0
53 52 1 1 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 1 0 0 0
57 52 1 1 0 0 0
56 58 1 0 0 0 0
55 59 1 0 0 0 0
57 60 1 0 0 0 0
52 61 1 0 0 0 0
53 51 1 0 0 0 0
62 63 1 0 0 0 0
46 62 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 69
M SMT 1 CH2OH
M SBV 1 69 -0.5584 -0.3025
S SKP 5
ID FL5FAAGA0026
FORMULA C39H50O24
EXACTMASS 902.269202528
AVERAGEMASS 902.7999
SMILES O(C(O7)C(C(C(O)C(CO)7)O)O)C(C(OC(C6=O)=C(Oc(c46)cc(OC(O5)C(C(O)C(O)C5C)O)cc4O)c(c3)ccc(c3)O)1)C(C(C(COC(O2)C(C(O)C(C2C)O)O)O1)O)O
M END