Mol:FL4DACGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0879 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0879 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5670 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5670 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0461 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0461 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0461 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0461 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5670 1.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5670 1.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0879 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0879 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5252 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5252 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0044 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0044 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0044 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0044 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5252 1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5252 1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5252 -0.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5252 -0.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5753 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5753 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 0.7816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 0.7816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6328 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6328 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6328 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6328 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5753 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5753 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5386 1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5386 1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4639 -0.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4639 -0.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1615 2.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1615 2.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6445 -1.2058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6445 -1.2058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1799 -1.5149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1799 -1.5149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0100 -0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0100 -0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1799 -0.3224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1799 -0.3224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6445 -0.0132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6445 -0.0132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8143 -0.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8143 -0.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4708 -0.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4708 -0.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7820 -1.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7820 -1.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0507 -1.9970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0507 -1.9970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5386 -1.2256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5386 -1.2256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5670 -0.7157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5670 -0.7157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2311 -1.8169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2311 -1.8169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1401 -2.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1401 -2.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6746 -2.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6746 -2.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1903 -2.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1903 -2.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8156 -1.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8156 -1.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3480 -1.9654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3480 -1.9654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7449 -2.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7449 -2.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4385 -2.3350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4385 -2.3350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9808 -2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9808 -2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0505 -1.1812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0505 -1.1812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7464 -1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7464 -1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 2 32 1 0 0 0 0 | + | 2 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 29 1 0 0 0 0 | + | 36 29 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 46 -6.1386 4.6834 | + | M SBV 1 46 -6.1386 4.6834 |
| − | S SKP 8 | + | S SKP 8 |
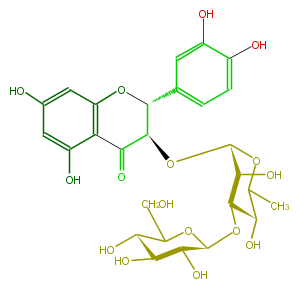
| − | ID FL4DACGS0023 | + | ID FL4DACGS0023 |
| − | KNApSAcK_ID C00008715 | + | KNApSAcK_ID C00008715 |
| − | NAME Huangqioside E | + | NAME Huangqioside E |
| − | CAS_RN 138822-69-2 | + | CAS_RN 138822-69-2 |
| − | FORMULA C27H32O16 | + | FORMULA C27H32O16 |
| − | EXACTMASS 612.1690349759999 | + | EXACTMASS 612.1690349759999 |
| − | AVERAGEMASS 612.53338 | + | AVERAGEMASS 612.53338 |
| − | SMILES C(C2c(c5)ccc(O)c5O)(OC(O4)C(C(C(O)C4C)OC(O3)C(O)C(O)C(O)C3CO)O)C(c(c1O2)c(O)cc(c1)O)=O | + | SMILES C(C2c(c5)ccc(O)c5O)(OC(O4)C(C(C(O)C4C)OC(O3)C(O)C(O)C(O)C3CO)O)C(c(c1O2)c(O)cc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.0879 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5670 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0461 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0461 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5670 1.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0879 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5252 -0.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0044 0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0044 0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5252 1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5252 -0.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5753 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 0.7816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6328 1.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6328 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5753 1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5386 1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4639 -0.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1615 2.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6445 -1.2058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1799 -1.5149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0100 -0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1799 -0.3224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6445 -0.0132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8143 -0.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4708 -0.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7820 -1.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0507 -1.9970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5386 -1.2256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5670 -0.7157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2311 -1.8169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1401 -2.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6746 -2.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1903 -2.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8156 -1.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3480 -1.9654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7449 -2.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4385 -2.3350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9808 -2.6131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0505 -1.1812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7464 -1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 19 1 0 0 0 0
2 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 29 1 0 0 0 0
38 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 46
M SMT 1 ^CH2OH
M SBV 1 46 -6.1386 4.6834
S SKP 8
ID FL4DACGS0023
KNApSAcK_ID C00008715
NAME Huangqioside E
CAS_RN 138822-69-2
FORMULA C27H32O16
EXACTMASS 612.1690349759999
AVERAGEMASS 612.53338
SMILES C(C2c(c5)ccc(O)c5O)(OC(O4)C(C(C(O)C4C)OC(O3)C(O)C(O)C(O)C3CO)O)C(c(c1O2)c(O)cc(c1)O)=O
M END