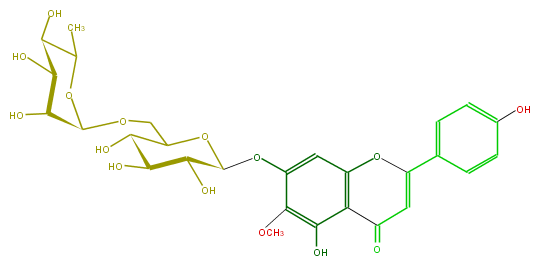
Mol:FL3FEAGS0039
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.6759 -0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6759 -0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3767 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3767 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3767 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3767 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6759 0.6565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6759 0.6565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9751 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9751 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9751 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9751 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9213 0.5663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9213 0.5663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2602 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2602 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5458 -0.5577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5458 -0.5577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8314 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8314 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8314 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8314 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5458 -2.2076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5458 -2.2076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2602 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2602 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1063 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1063 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3812 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3812 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3812 -1.8075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3812 -1.8075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1063 -2.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1063 -2.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5458 -2.7504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5458 -2.7504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1063 -2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1063 -2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3043 -0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3043 -0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3088 -0.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3088 -0.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8553 -0.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8553 -0.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9732 -0.6281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9732 -0.6281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0978 -0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0978 -0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5512 -0.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5512 -0.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4333 -0.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4333 -0.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8253 0.2280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8253 0.2280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4704 0.2820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4704 0.2820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5847 1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5847 1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4056 2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4056 2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0905 1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0905 1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2551 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2551 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4343 0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4343 0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7492 0.9009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7492 0.9009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5450 -1.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5450 -1.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7271 2.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7271 2.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1890 2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1890 2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8471 1.8164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8471 1.8164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9213 0.4201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9213 0.4201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8875 -0.4005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8875 -0.4005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5790 -0.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5790 -0.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1847 -2.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1847 -2.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9737 -1.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9737 -1.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 6 8 1 0 0 0 0 | + | 6 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 8 2 0 0 0 0 | + | 13 8 2 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 11 1 0 0 0 0 | + | 17 11 1 0 0 0 0 |
| − | 12 18 2 0 0 0 0 | + | 12 18 2 0 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 28 33 1 0 0 0 0 | + | 28 33 1 0 0 0 0 |
| − | 23 35 1 0 0 0 0 | + | 23 35 1 0 0 0 0 |
| − | 29 36 1 0 0 0 0 | + | 29 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 21 40 1 0 0 0 0 | + | 21 40 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 47 0.5659 0.5365 | + | M SBV 1 47 0.5659 0.5365 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0039 | + | ID FL3FEAGS0039 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES c(c(OC(C(O)4)OC(COC(O5)C(C(C(O)C(C)5)O)O)C(O)C(O)4)3)c(c(c(O)c3OC)2)OC(=CC2=O)c(c1)ccc(c1)O | + | SMILES c(c(OC(C(O)4)OC(COC(O5)C(C(C(O)C(C)5)O)O)C(O)C(O)4)3)c(c(c(O)c3OC)2)OC(=CC2=O)c(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
4.6759 -0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3767 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3767 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6759 0.6565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9751 0.2518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9751 -0.5574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9213 0.5663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2602 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5458 -0.5577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8314 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8314 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5458 -2.2076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2602 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1063 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3812 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3812 -1.8075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1063 -2.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5458 -2.7504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1063 -2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3043 -0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3088 -0.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8553 -0.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9732 -0.6281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0978 -0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5512 -0.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4333 -0.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8253 0.2280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4704 0.2820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5847 1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4056 2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0905 1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2551 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4343 0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7492 0.9009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5450 -1.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7271 2.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1890 2.8400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8471 1.8164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9213 0.4201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8875 -0.4005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5790 -0.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1847 -2.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9737 -1.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
6 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 8 2 0 0 0 0
10 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 11 1 0 0 0 0
12 18 2 0 0 0 0
17 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
20 24 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
28 33 1 0 0 0 0
23 35 1 0 0 0 0
29 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
21 40 1 0 0 0 0
22 41 1 0 0 0 0
42 43 1 0 0 0 0
16 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^OCH3
M SBV 1 47 0.5659 0.5365
S SKP 5
ID FL3FEAGS0039
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES c(c(OC(C(O)4)OC(COC(O5)C(C(C(O)C(C)5)O)O)C(O)C(O)4)3)c(c(c(O)c3OC)2)OC(=CC2=O)c(c1)ccc(c1)O
M END