Mol:FL3FEAGS0038
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.6818 1.6843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6818 1.6843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6818 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6818 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1328 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1328 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5839 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5839 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5839 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5839 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1328 1.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1328 1.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0349 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0349 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4860 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4860 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4860 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4860 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0349 1.9035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0349 1.9035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2066 0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2066 0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9369 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9369 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3966 1.6380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3966 1.6380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8564 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8564 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8564 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8564 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3966 2.6996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3966 2.6996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9369 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9369 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1328 0.3420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1328 0.3420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3741 -0.5829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.3741 -0.5829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.8585 -1.2636 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.8585 -1.2636 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1160 -0.9748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.1160 -0.9748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6005 -0.9671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.6005 -0.9671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0799 -0.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0799 -0.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6786 -0.7187 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.6786 -0.7187 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.1780 0.1488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1780 0.1488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6623 -1.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6623 -1.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3095 -1.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3095 -1.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0305 -2.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0305 -2.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2410 -1.5936 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.2410 -1.5936 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.7253 -2.2742 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7253 -2.2742 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9828 -1.9854 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9828 -1.9854 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2664 -1.9777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2664 -1.9777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7870 -1.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7870 -1.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5455 -1.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.5455 -1.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.2801 -2.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2801 -2.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5574 -2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5574 -2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9786 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9786 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8342 -0.5331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8342 -0.5331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3911 -0.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3911 -0.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3911 0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3911 0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7809 -0.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7809 -0.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4464 -0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4464 -0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0480 0.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0480 0.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3245 2.3030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3245 2.3030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1756 3.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1756 3.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3160 2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3160 2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0305 2.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0305 2.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0327 1.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0327 1.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0161 1.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0161 1.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 30 35 1 0 0 0 0 | + | 30 35 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 38 37 1 0 0 0 0 | + | 38 37 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 39 41 2 0 0 0 0 | + | 39 41 2 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 24 42 1 0 0 0 0 | + | 24 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 1 44 1 0 0 0 0 | + | 1 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 15 46 1 0 0 0 0 | + | 15 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 2 48 1 0 0 0 0 | + | 2 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 42 43 | + | M SAL 4 2 42 43 |
| − | M SBL 4 1 46 | + | M SBL 4 1 46 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 46 -0.4464 -0.1721 | + | M SVB 4 46 -0.4464 -0.1721 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 48 49 | + | M SAL 3 2 48 49 |
| − | M SBL 3 1 52 | + | M SBL 3 1 52 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 52 -0.0327 1.2542 | + | M SVB 3 52 -0.0327 1.2542 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 46 47 | + | M SAL 2 2 46 47 |
| − | M SBL 2 1 50 | + | M SBL 2 1 50 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 50 4.316 2.6996 | + | M SVB 2 50 4.316 2.6996 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 48 | + | M SBL 1 1 48 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 48 0.3245 2.303 | + | M SVB 1 48 0.3245 2.303 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FEAGS0038 | + | ID FL3FEAGS0038 |
| − | KNApSAcK_ID C00004496 | + | KNApSAcK_ID C00004496 |
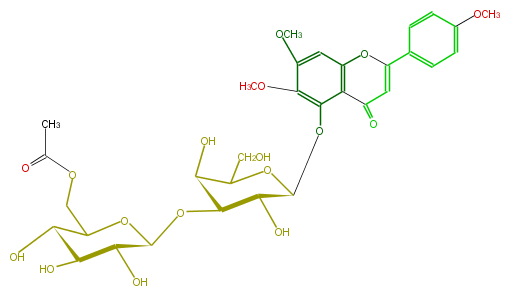
| − | NAME Scutellarein 6,7,4'-trimethyl ether 5-(6'''-acetylglucosyl)(1->3)-galactoside;5-[[3-O-(6-O-Acetyl-beta-D-glucopyranosyl)-beta-D-galactopyranosyl]oxy]-6,7-dimethoxy-2-(4-methoxyphenyl)-4H-1-benzopyran-4-one | + | NAME Scutellarein 6,7,4'-trimethyl ether 5-(6'''-acetylglucosyl)(1->3)-galactoside;5-[[3-O-(6-O-Acetyl-beta-D-glucopyranosyl)-beta-D-galactopyranosyl]oxy]-6,7-dimethoxy-2-(4-methoxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 158556-64-0 | + | CAS_RN 158556-64-0 |
| − | FORMULA C32H38O17 | + | FORMULA C32H38O17 |
| − | EXACTMASS 694.21089979 | + | EXACTMASS 694.21089979 |
| − | AVERAGEMASS 694.63392 | + | AVERAGEMASS 694.63392 |
| − | SMILES COc(c5)ccc(c5)C(O4)=CC(=O)c(c43)c(c(c(OC)c3)OC)O[C@@H]([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O[C@@H]([C@@H](O)2)OC([C@@H]([C@@H]2O)O)COC(C)=O | + | SMILES COc(c5)ccc(c5)C(O4)=CC(=O)c(c43)c(c(c(OC)c3)OC)O[C@@H]([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O[C@@H]([C@@H](O)2)OC([C@@H]([C@@H]2O)O)COC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
0.6818 1.6843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6818 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1328 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5839 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5839 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1328 1.9035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0349 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4860 1.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4860 1.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0349 1.9035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2066 0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9369 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3966 1.6380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8564 1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8564 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3966 2.6996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9369 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1328 0.3420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3741 -0.5829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.8585 -1.2636 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1160 -0.9748 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6005 -0.9671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0799 -0.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6786 -0.7187 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.1780 0.1488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6623 -1.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3095 -1.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0305 -2.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2410 -1.5936 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.7253 -2.2742 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9828 -1.9854 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2664 -1.9777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7870 -1.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5455 -1.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.2801 -2.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5574 -2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9786 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8342 -0.5331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3911 -0.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3911 0.4776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7809 -0.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4464 -0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0480 0.6267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3245 2.3030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1756 3.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3160 2.6996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0305 2.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0327 1.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0161 1.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
30 35 1 0 0 0 0
31 36 1 0 0 0 0
28 29 1 0 0 0 0
34 37 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
38 37 1 0 0 0 0
26 32 1 0 0 0 0
39 41 2 0 0 0 0
22 18 1 0 0 0 0
24 42 1 0 0 0 0
42 43 1 0 0 0 0
1 44 1 0 0 0 0
44 45 1 0 0 0 0
15 46 1 0 0 0 0
46 47 1 0 0 0 0
2 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 42 43
M SBL 4 1 46
M SMT 4 CH2OH
M SVB 4 46 -0.4464 -0.1721
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 48 49
M SBL 3 1 52
M SMT 3 OCH3
M SVB 3 52 -0.0327 1.2542
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 46 47
M SBL 2 1 50
M SMT 2 OCH3
M SVB 2 50 4.316 2.6996
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 48
M SMT 1 OCH3
M SVB 1 48 0.3245 2.303
S SKP 8
ID FL3FEAGS0038
KNApSAcK_ID C00004496
NAME Scutellarein 6,7,4'-trimethyl ether 5-(6'''-acetylglucosyl)(1->3)-galactoside;5-[[3-O-(6-O-Acetyl-beta-D-glucopyranosyl)-beta-D-galactopyranosyl]oxy]-6,7-dimethoxy-2-(4-methoxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 158556-64-0
FORMULA C32H38O17
EXACTMASS 694.21089979
AVERAGEMASS 694.63392
SMILES COc(c5)ccc(c5)C(O4)=CC(=O)c(c43)c(c(c(OC)c3)OC)O[C@@H]([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O[C@@H]([C@@H](O)2)OC([C@@H]([C@@H]2O)O)COC(C)=O
M END