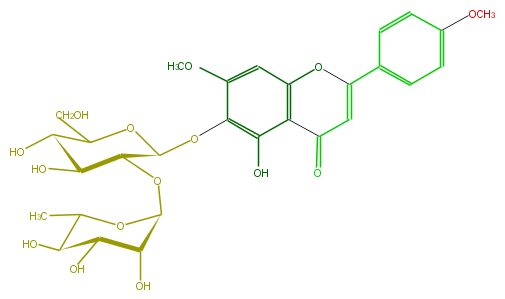
Mol:FL3FEAGS0037
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9400 1.4108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9400 1.4108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9400 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9400 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2781 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2781 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3840 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3840 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3840 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3840 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2781 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2781 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0461 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0461 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7080 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7080 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7080 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7080 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0461 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0461 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0312 -0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0312 -0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3698 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3698 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0446 1.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0446 1.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7193 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7193 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7193 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7193 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0446 2.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0446 2.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3698 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3698 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2781 -0.5593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2781 -0.5593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6532 0.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6532 0.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7464 0.2503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7464 0.2503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1410 -0.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1410 -0.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2692 -0.2098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2692 -0.2098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4279 -0.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4279 -0.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0392 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0392 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9298 0.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9298 0.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4426 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4426 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9597 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9597 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4588 -0.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4588 -0.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1484 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1484 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6035 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6035 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7283 -2.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7283 -2.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8476 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8476 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3924 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3924 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2678 -1.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2678 -1.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9359 -1.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9359 -1.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1661 -2.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1661 -2.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2895 -2.6568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2895 -2.6568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8476 -3.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8476 -3.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6487 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6487 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3827 3.0317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3827 3.0317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3939 2.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3939 2.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4426 2.2955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4426 2.2955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7054 0.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7054 0.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1897 1.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1897 1.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 1 39 1 0 0 0 0 | + | 1 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 15 41 1 0 0 0 0 | + | 15 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 25 43 1 0 0 0 0 | + | 25 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 44 0.7087 -0.3498 | + | M SBV 1 44 0.7087 -0.3498 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 46 -0.6746 -0.3895 | + | M SBV 2 46 -0.6746 -0.3895 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 43 44 | + | M SAL 3 2 43 44 |
| − | M SBL 3 1 48 | + | M SBL 3 1 48 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 48 0.7756 -0.6081 | + | M SBV 3 48 0.7756 -0.6081 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0037 | + | ID FL3FEAGS0037 |
| − | FORMULA C29H34O15 | + | FORMULA C29H34O15 |
| − | EXACTMASS 622.189770418 | + | EXACTMASS 622.189770418 |
| − | AVERAGEMASS 622.5712599999999 | + | AVERAGEMASS 622.5712599999999 |
| − | SMILES Oc(c(OC(C(OC(C5O)OC(C)C(C5O)O)4)OC(C(O)C4O)CO)3)c(C1=O)c(cc(OC)3)OC(c(c2)ccc(OC)c2)=C1 | + | SMILES Oc(c(OC(C(OC(C5O)OC(C)C(C5O)O)4)OC(C(O)C4O)CO)3)c(C1=O)c(cc(OC)3)OC(c(c2)ccc(OC)c2)=C1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.9400 1.4108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9400 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2781 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3840 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3840 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2781 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0461 0.2035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7080 0.5858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7080 1.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0461 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0312 -0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3698 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0446 1.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7193 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7193 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0446 2.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3698 2.5114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2781 -0.5593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6532 0.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7464 0.2503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1410 -0.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2692 -0.2098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4279 -0.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0392 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9298 0.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4426 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9597 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4588 -0.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1484 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6035 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7283 -2.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8476 -2.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3924 -1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2678 -1.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9359 -1.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1661 -2.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2895 -2.6568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8476 -3.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6487 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3827 3.0317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3939 2.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4426 2.2955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7054 0.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1897 1.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
2 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 28 1 0 0 0 0
23 19 1 0 0 0 0
39 40 1 0 0 0 0
1 39 1 0 0 0 0
41 42 1 0 0 0 0
15 41 1 0 0 0 0
43 44 1 0 0 0 0
25 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ OCH3
M SBV 1 44 0.7087 -0.3498
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 OCH3
M SBV 2 46 -0.6746 -0.3895
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 43 44
M SBL 3 1 48
M SMT 3 ^ CH2OH
M SBV 3 48 0.7756 -0.6081
S SKP 5
ID FL3FEAGS0037
FORMULA C29H34O15
EXACTMASS 622.189770418
AVERAGEMASS 622.5712599999999
SMILES Oc(c(OC(C(OC(C5O)OC(C)C(C5O)O)4)OC(C(O)C4O)CO)3)c(C1=O)c(cc(OC)3)OC(c(c2)ccc(OC)c2)=C1
M END