Mol:FL3FCACS0033
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.2334 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2334 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5189 -0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5189 -0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8044 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8044 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8044 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8044 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5189 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5189 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2334 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2334 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0900 -0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0900 -0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3755 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3755 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3755 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3755 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0900 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0900 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5189 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5189 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0348 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0348 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7492 -0.7789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7492 -0.7789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4637 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4637 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4637 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4637 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7492 0.8711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7492 0.8711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0348 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0348 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1471 0.8531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1471 0.8531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0900 -2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0900 -2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6850 -0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6850 -0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0481 -0.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0481 -0.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7792 2.4045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7792 2.4045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2422 1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2422 1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7046 1.0943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7046 1.0943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7542 0.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7542 0.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2914 0.8971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2914 0.8971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8290 1.5807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8290 1.5807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3294 1.9234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3294 1.9234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1630 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1630 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2422 2.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2422 2.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7950 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7950 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7913 1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7913 1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3787 0.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3787 0.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5803 0.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5803 0.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2132 0.3364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2132 0.3364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1994 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1994 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9978 0.8421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9978 0.8421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1548 1.4280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1548 1.4280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6346 1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6346 1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2745 0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2745 0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8273 0.6132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8273 0.6132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7518 -0.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7518 -0.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5032 2.5326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5032 2.5326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5375 2.2972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5375 2.2972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9981 2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9981 2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3613 2.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3613 2.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7626 1.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7626 1.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5864 1.5531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5864 1.5531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0094 2.2614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0094 2.2614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8344 2.2491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8344 2.2491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2362 1.5286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2362 1.5286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8131 0.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8131 0.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9882 0.8326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9882 0.8326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0600 1.5163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0600 1.5163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4211 2.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4211 2.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.1471 2.8078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.1471 2.8078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 7 25 1 0 0 0 0 | + | 7 25 1 0 0 0 0 |
| − | 31 24 1 0 0 0 0 | + | 31 24 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 50 55 1 0 0 0 0 | + | 50 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 43 28 1 0 0 0 0 | + | 43 28 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FCACS0033 | + | ID FL3FCACS0033 |
| − | KNApSAcK_ID C00014096 | + | KNApSAcK_ID C00014096 |
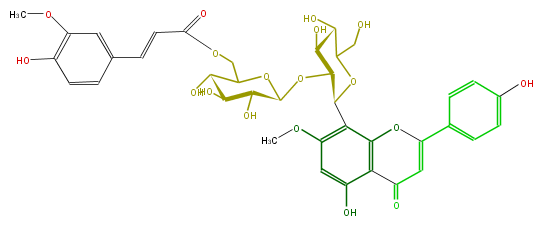
| − | NAME Isoswertisin 6'''-O-feruloyl 2''-O-glucoside | + | NAME Isoswertisin 6'''-O-feruloyl 2''-O-glucoside |
| − | CAS_RN 319480-85-8 | + | CAS_RN 319480-85-8 |
| − | FORMULA C38H40O18 | + | FORMULA C38H40O18 |
| − | EXACTMASS 784.2214644759999 | + | EXACTMASS 784.2214644759999 |
| − | AVERAGEMASS 784.7134 | + | AVERAGEMASS 784.7134 |
| − | SMILES c(c1)(ccc(C(=C3)Oc(c(C(O4)C(OC(C(O)5)OC(COC(C=Cc(c6)ccc(c(OC)6)O)=O)C(O)C5O)C(O)C(O)C(CO)4)2)c(C3=O)c(O)cc2OC)c1)O | + | SMILES c(c1)(ccc(C(=C3)Oc(c(C(O4)C(OC(C(O)5)OC(COC(C=Cc(c6)ccc(c(OC)6)O)=O)C(O)C5O)C(O)C(O)C(CO)4)2)c(C3=O)c(O)cc2OC)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
4.2334 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5189 -0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8044 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8044 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5189 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2334 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0900 -0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3755 -0.8291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3755 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0900 -2.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5189 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0348 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7492 -0.7789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4637 -0.3664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4637 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7492 0.8711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0348 0.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1471 0.8531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0900 -2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6850 -0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0481 -0.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7792 2.4045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2422 1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7046 1.0943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7542 0.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2914 0.8971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8290 1.5807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3294 1.9234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1630 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2422 2.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7950 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7913 1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3787 0.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5803 0.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2132 0.3364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1994 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9978 0.8421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1548 1.4280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6346 1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2745 0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8273 0.6132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7518 -0.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5032 2.5326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5375 2.2972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9981 2.8366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3613 2.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7626 1.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5864 1.5531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0094 2.2614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8344 2.2491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2362 1.5286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8131 0.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9882 0.8326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.0600 1.5163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.4211 2.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.1471 2.8078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
20 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
7 25 1 0 0 0 0
31 24 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 31 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
50 55 1 0 0 0 0
55 56 1 0 0 0 0
43 28 1 0 0 0 0
44 39 1 0 0 0 0
S SKP 8
ID FL3FCACS0033
KNApSAcK_ID C00014096
NAME Isoswertisin 6'''-O-feruloyl 2''-O-glucoside
CAS_RN 319480-85-8
FORMULA C38H40O18
EXACTMASS 784.2214644759999
AVERAGEMASS 784.7134
SMILES c(c1)(ccc(C(=C3)Oc(c(C(O4)C(OC(C(O)5)OC(COC(C=Cc(c6)ccc(c(OC)6)O)=O)C(O)C5O)C(O)C(O)C(CO)4)2)c(C3=O)c(O)cc2OC)c1)O
M END