Mol:FL3FADGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6137 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6137 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6137 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6137 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0874 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0874 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7883 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7883 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7883 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7883 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0874 -0.4751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0874 -0.4751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4894 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4894 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1905 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1905 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1905 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1905 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4894 -0.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4894 -0.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4894 -2.8208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4894 -2.8208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8913 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8913 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 -0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 -0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3203 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3203 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3203 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3203 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8913 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8913 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0874 -2.9019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0874 -2.9019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9697 0.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9697 0.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3133 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3133 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2358 -0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2358 -0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6748 -1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6748 -1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8670 -0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8670 -0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0875 -0.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0875 -0.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6540 -0.3799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6540 -0.3799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3606 -0.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3606 -0.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9697 -0.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9697 -0.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4448 -1.2098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4448 -1.2098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1553 -1.4445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1553 -1.4445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2095 2.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2095 2.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9963 -0.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9963 -0.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6132 -0.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6132 -0.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6994 0.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6994 0.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 34 0.0000 -0.7073 | + | M SBV 1 34 0.0000 -0.7073 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 32 33 34 | + | M SAL 2 3 32 33 34 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 ^ COOH | + | M SMT 2 ^ COOH |
| − | M SBV 2 37 0.6357 -0.5385 | + | M SBV 2 37 0.6357 -0.5385 |
| − | S SKP 5 | + | S SKP 5 |
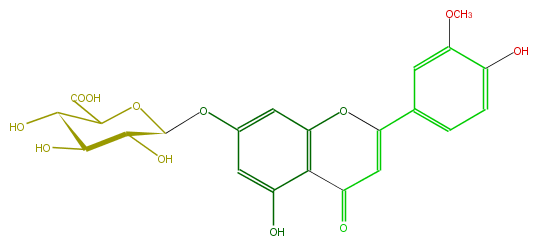
| − | ID FL3FADGS0005 | + | ID FL3FADGS0005 |
| − | FORMULA C22H20O12 | + | FORMULA C22H20O12 |
| − | EXACTMASS 476.095476104 | + | EXACTMASS 476.095476104 |
| − | AVERAGEMASS 476.387 | + | AVERAGEMASS 476.387 |
| − | SMILES c(c4O)(cc(cc4)C(O3)=CC(c(c23)c(cc(c2)OC(O1)C(O)C(C(O)C1C(O)=O)O)O)=O)OC | + | SMILES c(c4O)(cc(cc4)C(O3)=CC(c(c23)c(cc(c2)OC(O1)C(O)C(C(O)C1C(O)=O)O)O)=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-0.6137 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6137 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0874 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7883 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7883 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0874 -0.4751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4894 -2.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1905 -1.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1905 -0.8798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4894 -0.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4894 -2.8208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8913 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 -0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3203 -0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3203 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8913 0.3499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0874 -2.9019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9697 0.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3133 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2358 -0.5285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6748 -1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8670 -0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0875 -0.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6540 -0.3799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3606 -0.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9697 -0.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4448 -1.2098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1553 -1.4445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2095 2.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9963 -0.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6132 -0.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6994 0.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
30 31 1 0 0 0 0
16 30 1 0 0 0 0
32 34 1 0 0 0 0
32 33 2 0 0 0 0
26 32 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 34
M SMT 1 OCH3
M SBV 1 34 0.0000 -0.7073
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 32 33 34
M SBL 2 1 37
M SMT 2 ^ COOH
M SBV 2 37 0.6357 -0.5385
S SKP 5
ID FL3FADGS0005
FORMULA C22H20O12
EXACTMASS 476.095476104
AVERAGEMASS 476.387
SMILES c(c4O)(cc(cc4)C(O3)=CC(c(c23)c(cc(c2)OC(O1)C(O)C(C(O)C1C(O)=O)O)O)=O)OC
M END