Mol:FL3FACDS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8657 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8657 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8657 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8657 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1512 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1512 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4368 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4368 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4368 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4368 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1512 0.4524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1512 0.4524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2777 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2777 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9922 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9922 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9922 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9922 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2777 0.4524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2777 0.4524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2777 -1.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2777 -1.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5799 0.4523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5799 0.4523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0217 3.4051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0217 3.4051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7994 2.8159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7994 2.8159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4695 1.9675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4695 1.9675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4606 1.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4606 1.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8656 1.7436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8656 1.7436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2575 2.4859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2575 2.4859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3907 4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3907 4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7994 3.6208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7994 3.6208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8850 1.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8850 1.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1512 -2.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1512 -2.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7857 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7857 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5386 0.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5386 0.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2916 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2916 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2916 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2916 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5386 1.7828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5386 1.7828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7857 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7857 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0439 1.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0439 1.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0439 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0439 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5595 -1.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5595 -1.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0828 -1.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0828 -1.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3963 -1.5712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3963 -1.5712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7340 -1.5641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7340 -1.5641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2153 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2153 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8159 -1.3996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8159 -1.3996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1179 -1.5313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1179 -1.5313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7148 -1.8382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7148 -1.8382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9903 -1.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9903 -1.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6963 -1.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6963 -1.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9857 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9857 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2112 -1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2112 -1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9857 -2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9857 -2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6963 -2.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6963 -2.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4709 -2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4709 -2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4921 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4921 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1179 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1179 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6963 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6963 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2714 -3.4609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2714 -3.4609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6880 -4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6880 -4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2903 -0.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2903 -0.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0395 -0.1761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0395 -0.1761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7995 2.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7995 2.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1181 2.4141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1181 2.4141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 9 1 0 0 0 0 | + | 23 9 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 2 1 0 0 0 0 | + | 34 2 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 41 30 1 0 0 0 0 | + | 41 30 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 36 51 1 0 0 0 0 | + | 36 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 18 53 1 0 0 0 0 | + | 18 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 54 -0.2857 1.0663 | + | M SBV 1 54 -0.2857 1.0663 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 57 0.4744 -0.4744 | + | M SBV 2 57 0.4744 -0.4744 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 53 54 | + | M SAL 3 2 53 54 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 59 -0.4579 -0.4579 | + | M SBV 3 59 -0.4579 -0.4579 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0024 | + | ID FL3FACDS0024 |
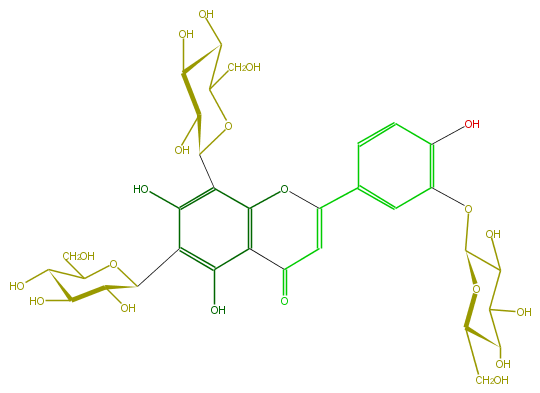
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES c(c1C(C(O)6)OC(C(O)C6O)CO)(O3)c(C(C=C(c(c4)cc(OC(O5)C(C(O)C(O)C5CO)O)c(O)c4)3)=O)c(c(C(O2)C(O)C(O)C(O)C2CO)c1O)O | + | SMILES c(c1C(C(O)6)OC(C(O)C6O)CO)(O3)c(C(C=C(c(c4)cc(OC(O5)C(C(O)C(O)C5CO)O)c(O)c4)3)=O)c(c(C(O2)C(O)C(O)C(O)C2CO)c1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.8657 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8657 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1512 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4368 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4368 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1512 0.4524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2777 -1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9922 -0.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9922 0.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2777 0.4524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2777 -1.8407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5799 0.4523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0217 3.4051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7994 2.8159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4695 1.9675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4606 1.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8656 1.7436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2575 2.4859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3907 4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7994 3.6208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8850 1.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1512 -2.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7857 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5386 0.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2916 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2916 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5386 1.7828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7857 1.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0439 1.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0439 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5595 -1.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0828 -1.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3963 -1.5712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7340 -1.5641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2153 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8159 -1.3996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1179 -1.5313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7148 -1.8382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9903 -1.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6963 -1.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9857 -0.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2112 -1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9857 -2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6963 -2.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4709 -2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4921 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1179 -2.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6963 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2714 -3.4609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6880 -4.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2903 -0.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0395 -0.1761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7995 2.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1181 2.4141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
23 9 1 0 0 0 0
26 29 1 0 0 0 0
25 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 2 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
41 30 1 0 0 0 0
43 49 1 0 0 0 0
49 50 1 0 0 0 0
51 52 1 0 0 0 0
36 51 1 0 0 0 0
53 54 1 0 0 0 0
18 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 54
M SMT 1 CH2OH
M SBV 1 54 -0.2857 1.0663
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 57
M SMT 2 ^ CH2OH
M SBV 2 57 0.4744 -0.4744
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 53 54
M SBL 3 1 59
M SMT 3 CH2OH
M SBV 3 59 -0.4579 -0.4579
S SKP 5
ID FL3FACDS0024
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES c(c1C(C(O)6)OC(C(O)C6O)CO)(O3)c(C(C=C(c(c4)cc(OC(O5)C(C(O)C(O)C5CO)O)c(O)c4)3)=O)c(c(C(O2)C(O)C(O)C(O)C2CO)c1O)O
M END