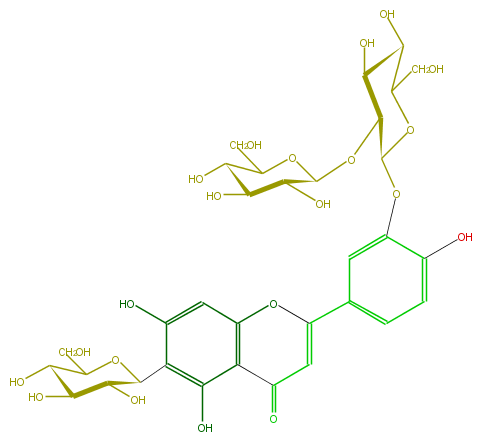
Mol:FL3FACDS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4993 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4993 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4993 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4993 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7849 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7849 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0704 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0704 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0704 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0704 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7849 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7849 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6441 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6441 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3585 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3585 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3585 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3585 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6441 -1.6614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6441 -1.6614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6441 -3.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6441 -3.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2135 -1.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2135 -1.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7849 -4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7849 -4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1522 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1522 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9051 -2.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9051 -2.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6579 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6579 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6579 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6579 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9051 -0.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9051 -0.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1522 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1522 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4104 -0.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4104 -0.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8457 -2.9045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8457 -2.9045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3690 -3.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3690 -3.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6825 -3.2668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6825 -3.2668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0201 -3.2596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0201 -3.2596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5014 -2.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5014 -2.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1020 -3.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1020 -3.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4104 -3.2306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4104 -3.2306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0303 -3.5338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0303 -3.5338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1374 -3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1374 -3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2329 3.4811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2329 3.4811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4551 2.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4551 2.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7851 2.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7851 2.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7939 1.2248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7939 1.2248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3890 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3890 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9972 2.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9972 2.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8548 4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8548 4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4551 3.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4551 3.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2005 1.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2005 1.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3192 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3192 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1575 0.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1575 0.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8440 0.7642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8440 0.7642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5064 0.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5064 0.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0250 1.2527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0250 1.2527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4245 0.9358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4245 0.9358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8308 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8308 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4629 0.4972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4629 0.4972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5772 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5772 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1080 0.5412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1080 0.5412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1609 1.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1609 1.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8626 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8626 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4760 3.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4760 3.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3936 2.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3936 2.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5800 -2.6171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5800 -2.6171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 -2.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 -2.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 39 45 1 0 0 0 0 | + | 39 45 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 38 1 0 0 0 0 | + | 42 38 1 0 0 0 0 |
| − | 18 48 1 0 0 0 0 | + | 18 48 1 0 0 0 0 |
| − | 48 33 1 0 0 0 0 | + | 48 33 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 44 49 1 0 0 0 0 | + | 44 49 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 35 51 1 0 0 0 0 | + | 35 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 26 53 1 0 0 0 0 | + | 26 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 55 0.5854 -0.5854 | + | M SBV 1 55 0.5854 -0.5854 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 57 -0.4788 -0.4788 | + | M SBV 2 57 -0.4788 -0.4788 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 53 54 | + | M SAL 3 2 53 54 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 ^CH2OH | + | M SMT 3 ^CH2OH |
| − | M SBV 3 59 0.4781 -0.4781 | + | M SBV 3 59 0.4781 -0.4781 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0015 | + | ID FL3FACDS0015 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES C(O)(C6O)C(OC(C6O)OC(C(O)5)C(OC(CO)C(O)5)Oc(c4O)cc(cc4)C(O3)=CC(c(c31)c(c(C(C(O)2)OC(CO)C(O)C(O)2)c(c1)O)O)=O)CO | + | SMILES C(O)(C6O)C(OC(C6O)OC(C(O)5)C(OC(CO)C(O)5)Oc(c4O)cc(cc4)C(O3)=CC(c(c31)c(c(C(C(O)2)OC(CO)C(O)C(O)2)c(c1)O)O)=O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.4993 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4993 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7849 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0704 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0704 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7849 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6441 -3.3114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3585 -2.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3585 -2.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6441 -1.6614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6441 -3.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2135 -1.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7849 -4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1522 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9051 -2.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6579 -1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6579 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9051 -0.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1522 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4104 -0.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8457 -2.9045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3690 -3.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6825 -3.2668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0201 -3.2596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5014 -2.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1020 -3.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4104 -3.2306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0303 -3.5338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1374 -3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2329 3.4811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4551 2.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7851 2.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7939 1.2248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3890 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9972 2.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8548 4.1360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4551 3.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2005 1.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3192 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1575 0.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8440 0.7642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5064 0.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0250 1.2527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4245 0.9358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8308 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4629 0.4972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5772 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1080 0.5412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1609 1.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8626 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4760 3.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3936 2.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5800 -2.6171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 -2.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
14 9 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
39 40 1 1 0 0 0
40 41 1 1 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 39 1 0 0 0 0
39 45 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 38 1 0 0 0 0
18 48 1 0 0 0 0
48 33 1 0 0 0 0
49 50 1 0 0 0 0
44 49 1 0 0 0 0
51 52 1 0 0 0 0
35 51 1 0 0 0 0
53 54 1 0 0 0 0
26 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 55
M SMT 1 ^CH2OH
M SBV 1 55 0.5854 -0.5854
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 57
M SMT 2 CH2OH
M SBV 2 57 -0.4788 -0.4788
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 53 54
M SBL 3 1 59
M SMT 3 ^CH2OH
M SBV 3 59 0.4781 -0.4781
S SKP 5
ID FL3FACDS0015
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES C(O)(C6O)C(OC(C6O)OC(C(O)5)C(OC(CO)C(O)5)Oc(c4O)cc(cc4)C(O3)=CC(c(c31)c(c(C(C(O)2)OC(CO)C(O)C(O)2)c(c1)O)O)=O)CO
M END