Mol:FL3FACDS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7270 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7270 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7270 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7270 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1707 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1707 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3856 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3856 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3856 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3856 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1707 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1707 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9419 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9419 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4982 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4982 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4982 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4982 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9419 -0.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9419 -0.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9419 -2.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9419 -2.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2831 -0.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2831 -0.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1162 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1162 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7024 -0.8956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7024 -0.8956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2886 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2886 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2886 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2886 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7024 0.4582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7024 0.4582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1162 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1162 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8745 0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8745 0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0541 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0541 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5515 1.4418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5515 1.4418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2946 0.7812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2946 0.7812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2877 0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2877 0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1757 0.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1757 0.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1294 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1294 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1077 2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1077 2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5863 2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5863 2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9300 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9300 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8745 -0.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8745 -0.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1707 -2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1707 -2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3607 -0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3607 -0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9895 -1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9895 -1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4550 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4550 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9392 -0.9721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9392 -0.9721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3140 -0.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3140 -0.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7816 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7816 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8745 -0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8745 -0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5681 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5681 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1487 -1.4917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1487 -1.4917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1801 -0.0598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1801 -0.0598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3832 -0.2733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3832 -0.2733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3859 1.4824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3859 1.4824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1004 1.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1004 1.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 12 1 0 0 0 0 | + | 34 12 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 44 -6.7725 6.1966 | + | M SBV 1 44 -6.7725 6.1966 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -5.8588 5.7100 | + | M SBV 2 46 -5.8588 5.7100 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACDS0003 | + | ID FL3FACDS0003 |
| − | KNApSAcK_ID C00006202 | + | KNApSAcK_ID C00006202 |
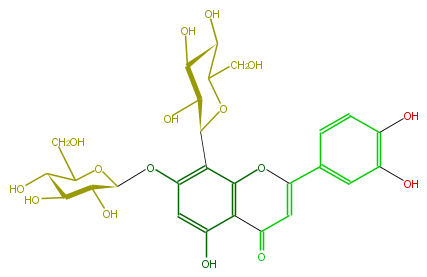
| − | NAME Orientin 7-glucoside | + | NAME Orientin 7-glucoside |
| − | CAS_RN 61361-16-8 | + | CAS_RN 61361-16-8 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(C1O)(C(O)C(c(c42)c(OC(C5O)OC(C(O)C5O)CO)cc(O)c2C(C=C(O4)c(c3)ccc(O)c3O)=O)OC1CO)O | + | SMILES C(C1O)(C(O)C(c(c42)c(OC(C5O)OC(C(O)C5O)CO)cc(O)c2C(C=C(O4)c(c3)ccc(O)c3O)=O)OC1CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.7270 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7270 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1707 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3856 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3856 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1707 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9419 -1.8623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4982 -1.5412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4982 -0.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9419 -0.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9419 -2.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2831 -0.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1162 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7024 -0.8956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2886 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2886 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7024 0.4582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1162 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8745 0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0541 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5515 1.4418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2946 0.7812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2877 0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1757 0.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1294 1.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1077 2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5863 2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9300 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8745 -0.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1707 -2.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3607 -0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9895 -1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4550 -0.9776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9392 -0.9721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3140 -0.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7816 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8745 -0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5681 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1487 -1.4917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1801 -0.0598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3832 -0.2733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3859 1.4824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1004 1.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
15 29 1 0 0 0 0
3 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 12 1 0 0 0 0
36 40 1 0 0 0 0
40 41 1 0 0 0 0
25 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 ^CH2OH
M SBV 1 44 -6.7725 6.1966
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -5.8588 5.7100
S SKP 8
ID FL3FACDS0003
KNApSAcK_ID C00006202
NAME Orientin 7-glucoside
CAS_RN 61361-16-8
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(C1O)(C(O)C(c(c42)c(OC(C5O)OC(C(O)C5O)CO)cc(O)c2C(C=C(O4)c(c3)ccc(O)c3O)=O)OC1CO)O
M END