Mol:FL3FACCS0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1935 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1935 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1935 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1935 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4892 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4892 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2151 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2151 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2151 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2151 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4892 0.7549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4892 0.7549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9195 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9195 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6238 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6238 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6238 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6238 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9195 0.7549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9195 0.7549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9195 -1.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9195 -1.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4892 -1.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4892 -1.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4845 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4845 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2267 0.4568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2267 0.4568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9689 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9689 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9689 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9689 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2267 2.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2267 2.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4845 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4845 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7107 2.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7107 2.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8598 0.7329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8598 0.7329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0746 -0.4047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0746 -0.4047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4218 -1.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4218 -1.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7690 -0.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7690 -0.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8811 -0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8811 -0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5078 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5078 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2129 -0.6136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2129 -0.6136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7107 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7107 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1884 -0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1884 -0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0182 -1.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0182 -1.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8619 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8619 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6869 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6869 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1084 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1084 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2845 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2845 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4184 -2.5837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4184 -2.5837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6869 -2.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6869 -2.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8619 -1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8619 -1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2267 3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2267 3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8755 0.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8755 0.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5466 0.8491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5466 0.8491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1084 -1.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1084 -1.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2843 -2.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2843 -2.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 17 37 1 0 0 0 0 | + | 17 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 26 38 1 0 0 0 0 | + | 26 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 43 0.6626 -0.6626 | + | M SBV 1 43 0.6626 -0.6626 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 45 0.0000 -0.7738 | + | M SBV 2 45 0.0000 -0.7738 |
| − | S SKP 5 | + | S SKP 5 |
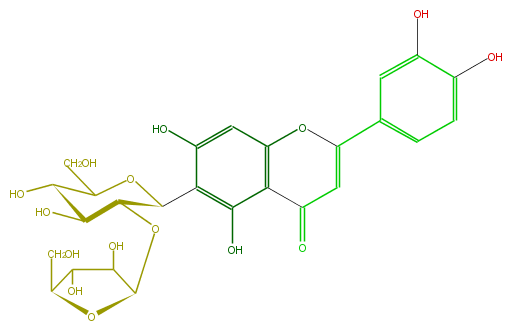
| − | ID FL3FACCS0017 | + | ID FL3FACCS0017 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES C(O)C(C(O)5)OC(C(O)5)OC(C1O)C(c(c2O)c(cc(O3)c(C(C=C(c(c4)ccc(c4O)O)3)=O)2)O)OC(C1O)CO | + | SMILES C(O)C(C(O)5)OC(C(O)5)OC(C1O)C(c(c2O)c(cc(O3)c(C(C=C(c(c4)ccc(c4O)O)3)=O)2)O)OC(C1O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.1935 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1935 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4892 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2151 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2151 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4892 0.7549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9195 -0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6238 -0.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6238 0.3483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9195 0.7549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9195 -1.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4892 -1.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4845 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2267 0.4568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9689 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9689 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2267 2.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4845 1.7424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7107 2.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8598 0.7329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0746 -0.4047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4218 -1.1359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7690 -0.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8811 -0.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5078 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2129 -0.6136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7107 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1884 -0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0182 -1.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8619 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6869 -2.0978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1084 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2845 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4184 -2.5837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6869 -2.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8619 -1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2267 3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8755 0.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5466 0.8491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1084 -1.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2843 -2.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 30 1 0 0 0 0
31 35 1 0 0 0 0
30 36 1 0 0 0 0
34 29 1 0 0 0 0
17 37 1 0 0 0 0
38 39 1 0 0 0 0
26 38 1 0 0 0 0
40 41 1 0 0 0 0
32 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 ^ CH2OH
M SBV 1 43 0.6626 -0.6626
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 CH2OH
M SBV 2 45 0.0000 -0.7738
S SKP 5
ID FL3FACCS0017
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES C(O)C(C(O)5)OC(C(O)5)OC(C1O)C(c(c2O)c(cc(O3)c(C(C=C(c(c4)ccc(c4O)O)3)=O)2)O)OC(C1O)CO
M END