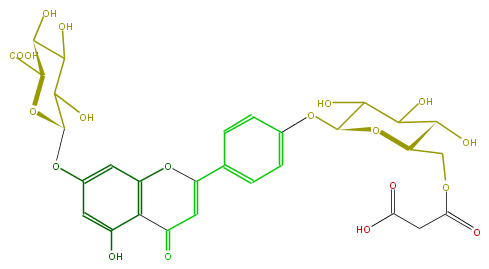
Mol:FL3FAAGS0055
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3495 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3495 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3495 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3495 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6929 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6929 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0362 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0362 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0362 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0362 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6929 -0.7428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6929 -0.7428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3797 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3797 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7230 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7230 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7230 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7230 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3797 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3797 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3797 -2.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3797 -2.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0667 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0667 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6025 -1.1293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6025 -1.1293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2717 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2717 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2717 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2717 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6025 0.4162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6025 0.4162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0667 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0667 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0059 -0.7430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0059 -0.7430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9541 0.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9541 0.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6929 -2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6929 -2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2213 0.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2213 0.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5830 0.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5830 0.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4856 0.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4856 0.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2651 -0.3560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2651 -0.3560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9037 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9037 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0008 0.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0008 0.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3534 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3534 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5550 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5550 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5880 -0.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5880 -0.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0429 -0.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0429 -0.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1254 -1.2203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1254 -1.2203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1265 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1265 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8456 -2.2720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8456 -2.2720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5074 -2.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5074 -2.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8881 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8881 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8881 -1.1740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8881 -1.1740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2688 -2.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2688 -2.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5358 0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5358 0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8041 0.1272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8041 0.1272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0364 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0364 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8041 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8041 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5358 2.1791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5358 2.1791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3037 1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3037 1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2327 2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2327 2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8646 2.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8646 2.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3558 0.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3558 0.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0136 1.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0136 1.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0136 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0136 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8456 1.3722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8456 1.3722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 19 22 1 0 0 0 0 | + | 19 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 38 1 1 0 0 0 | + | 43 38 1 1 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 41 45 1 0 0 0 0 | + | 41 45 1 0 0 0 0 |
| − | 18 39 1 0 0 0 0 | + | 18 39 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 43 47 1 0 0 0 0 | + | 43 47 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 47 48 49 | + | M SAL 1 3 47 48 49 |
| − | M SBL 1 1 53 | + | M SBL 1 1 53 |
| − | M SMT 1 ^COOH | + | M SMT 1 ^COOH |
| − | M SBV 1 53 0.7098 -0.4858 | + | M SBV 1 53 0.7098 -0.4858 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAAGS0055 | + | ID FL3FAAGS0055 |
| − | FORMULA C30H30O19 | + | FORMULA C30H30O19 |
| − | EXACTMASS 694.138128778 | + | EXACTMASS 694.138128778 |
| − | AVERAGEMASS 694.5478 | + | AVERAGEMASS 694.5478 |
| − | SMILES O(C(C(O)5)OC(C(O)C(O)5)C(O)=O)c(c4)cc(c(c(O)4)3)OC(=CC(=O)3)c(c1)ccc(OC(O2)C(C(C(C2COC(=O)CC(O)=O)O)O)O)c1 | + | SMILES O(C(C(O)5)OC(C(O)C(O)5)C(O)=O)c(c4)cc(c(c(O)4)3)OC(=CC(=O)3)c(c1)ccc(OC(O2)C(C(C(C2COC(=O)CC(O)=O)O)O)O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-3.3495 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3495 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6929 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0362 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0362 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6929 -0.7428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3797 -2.2592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7230 -1.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7230 -1.1219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3797 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3797 -2.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0667 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6025 -1.1293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2717 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2717 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6025 0.4162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0667 0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0059 -0.7430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9541 0.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6929 -2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2213 0.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5830 0.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4856 0.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2651 -0.3560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9037 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0008 0.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3534 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5550 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5880 -0.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0429 -0.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1254 -1.2203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1265 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8456 -2.2720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5074 -2.2144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8881 -1.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8881 -1.1740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2688 -2.2144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5358 0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8041 0.1272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0364 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8041 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5358 2.1791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3037 1.3668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2327 2.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8646 2.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3558 0.4191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0136 1.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0136 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8456 1.3722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
19 22 1 0 0 0 0
23 24 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 1 0 0 0
43 38 1 1 0 0 0
42 44 1 0 0 0 0
41 45 1 0 0 0 0
18 39 1 0 0 0 0
40 46 1 0 0 0 0
47 48 2 0 0 0 0
47 49 1 0 0 0 0
43 47 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 47 48 49
M SBL 1 1 53
M SMT 1 ^COOH
M SBV 1 53 0.7098 -0.4858
S SKP 5
ID FL3FAAGS0055
FORMULA C30H30O19
EXACTMASS 694.138128778
AVERAGEMASS 694.5478
SMILES O(C(C(O)5)OC(C(O)C(O)5)C(O)=O)c(c4)cc(c(c(O)4)3)OC(=CC(=O)3)c(c1)ccc(OC(O2)C(C(C(C2COC(=O)CC(O)=O)O)O)O)c1
M END