Mol:FL3FAADS0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1799 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1799 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1799 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1799 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3764 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3764 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9327 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9327 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9327 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9327 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3764 -0.6810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3764 -0.6810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4890 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4890 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0453 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0453 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0453 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0453 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4890 -0.6810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4890 -0.6810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4890 -2.4665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4890 -2.4665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7360 -0.6811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7360 -0.6811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6012 1.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6012 1.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0044 1.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0044 1.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2525 0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2525 0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2594 0.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2594 0.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7227 0.5036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7227 0.5036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4177 1.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4177 1.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1332 2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1332 2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0392 2.0536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0392 2.0536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3829 0.2993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3829 0.2993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3764 -2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3764 -2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7252 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7252 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3114 -0.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3114 -0.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8976 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8976 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8976 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8976 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3114 0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3114 0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7252 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7252 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4835 0.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4835 0.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9646 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9646 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6609 1.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6609 1.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6665 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6665 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9631 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9631 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5523 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5523 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8742 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8742 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5180 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5180 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8400 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8400 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5180 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5180 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8742 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8742 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4835 1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4835 1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7930 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7930 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4218 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4218 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8873 -1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8873 -1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3715 -1.1992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3715 -1.1992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7463 -0.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7463 -0.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2139 -1.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2139 -1.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3068 -1.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3068 -1.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0004 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0004 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5810 -1.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5810 -1.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6124 -0.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6124 -0.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8155 -0.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8155 -0.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9930 1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9930 1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7074 1.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7074 1.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 9 23 1 0 0 0 0 | + | 9 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 21 1 0 0 0 0 | + | 30 21 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 12 1 0 0 0 0 | + | 44 12 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 18 52 1 0 0 0 0 | + | 18 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 55 -5.6273 6.3820 | + | M SBV 1 55 -5.6273 6.3820 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 57 -4.6536 5.9300 | + | M SBV 2 57 -4.6536 5.9300 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAADS0018 | + | ID FL3FAADS0018 |
| − | KNApSAcK_ID C00006325 | + | KNApSAcK_ID C00006325 |
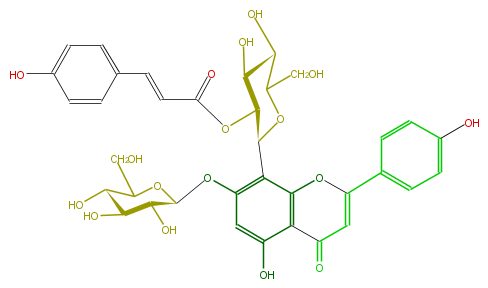
| − | NAME Vitexin 7-O-glucoside 2''-p-coumarate | + | NAME Vitexin 7-O-glucoside 2''-p-coumarate |
| − | CAS_RN 93446-17-4 | + | CAS_RN 93446-17-4 |
| − | FORMULA C36H36O17 | + | FORMULA C36H36O17 |
| − | EXACTMASS 740.1952497259999 | + | EXACTMASS 740.1952497259999 |
| − | AVERAGEMASS 740.66084 | + | AVERAGEMASS 740.66084 |
| − | SMILES C(C1=O)=C(c(c6)ccc(O)c6)Oc(c(C(C(OC(C=Cc(c5)ccc(c5)O)=O)4)OC(C(O)C(O)4)CO)2)c(c(cc2OC(C(O)3)OC(CO)C(O)C(O)3)O)1 | + | SMILES C(C1=O)=C(c(c6)ccc(O)c6)Oc(c(C(C(OC(C=Cc(c5)ccc(c5)O)=O)4)OC(C(O)C(O)4)CO)2)c(c(cc2OC(C(O)3)OC(CO)C(O)C(O)3)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-0.1799 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1799 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3764 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9327 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9327 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3764 -0.6810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4890 -1.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0453 -1.6445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0453 -1.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4890 -0.6810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4890 -2.4665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7360 -0.6811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6012 1.7972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0044 1.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2525 0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2594 0.0404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7227 0.5036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4177 1.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1332 2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0392 2.0536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3829 0.2993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3764 -2.6078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7252 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3114 -0.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8976 -0.5780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8976 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3114 0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7252 0.0989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4835 0.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9646 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6609 1.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6665 0.8809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9631 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5523 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8742 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5180 1.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8400 1.3947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5180 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8742 0.8371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4835 1.3947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7930 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4218 -1.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8873 -1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3715 -1.1992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7463 -0.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2139 -1.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3068 -1.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0004 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5810 -1.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6124 -0.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8155 -0.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9930 1.4137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7074 1.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
9 23 1 0 0 0 0
26 29 1 0 0 0 0
30 21 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 12 1 0 0 0 0
46 50 1 0 0 0 0
50 51 1 0 0 0 0
18 52 1 0 0 0 0
52 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 55
M SMT 1 ^CH2OH
M SBV 1 55 -5.6273 6.3820
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 57
M SMT 2 CH2OH
M SBV 2 57 -4.6536 5.9300
S SKP 8
ID FL3FAADS0018
KNApSAcK_ID C00006325
NAME Vitexin 7-O-glucoside 2''-p-coumarate
CAS_RN 93446-17-4
FORMULA C36H36O17
EXACTMASS 740.1952497259999
AVERAGEMASS 740.66084
SMILES C(C1=O)=C(c(c6)ccc(O)c6)Oc(c(C(C(OC(C=Cc(c5)ccc(c5)O)=O)4)OC(C(O)C(O)4)CO)2)c(c(cc2OC(C(O)3)OC(CO)C(O)C(O)3)O)1
M END