Mol:FL2FBANC0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7383 -0.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7383 -0.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0241 -0.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0241 -0.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7383 -1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7383 -1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0232 -2.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0232 -2.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6911 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6911 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6906 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6906 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4058 -2.0676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4058 -2.0676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1200 -1.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1200 -1.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1195 -0.8297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1195 -0.8297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4048 -0.4176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4048 -0.4176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4061 -2.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4061 -2.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0227 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0227 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6415 -3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6415 -3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7748 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7748 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4893 -0.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4893 -0.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2037 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2037 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2037 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2037 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4893 0.7861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4893 0.7861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7748 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7748 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7969 0.7161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7969 0.7161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0241 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0241 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6904 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6904 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6904 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6904 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0241 2.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0241 2.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7386 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7386 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7386 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7386 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3981 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3981 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1125 1.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1125 1.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8270 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8270 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8270 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8270 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1125 3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1125 3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3981 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3981 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4092 3.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4092 3.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3934 1.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3934 1.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0684 1.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0684 1.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7577 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7577 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7577 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7577 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4721 3.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4721 3.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1866 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1866 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1866 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1866 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4721 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4721 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7969 3.1550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7969 3.1550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3844 -0.4583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3844 -0.4583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 4 12 1 0 0 0 0 | + | 4 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 9 14 1 0 0 0 0 | + | 9 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 2 21 1 6 0 0 0 | + | 2 21 1 6 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 23 27 1 6 0 0 0 | + | 23 27 1 6 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 25 34 1 6 0 0 0 | + | 25 34 1 6 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 1 43 1 0 0 0 0 | + | 1 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FBANC0005 | + | ID FL2FBANC0005 |
| − | KNApSAcK_ID C00014289 | + | KNApSAcK_ID C00014289 |
| − | NAME Calyxin K | + | NAME Calyxin K |
| − | CAS_RN 332877-78-8 | + | CAS_RN 332877-78-8 |
| − | FORMULA C35H34O8 | + | FORMULA C35H34O8 |
| − | EXACTMASS 582.225368064 | + | EXACTMASS 582.225368064 |
| − | AVERAGEMASS 582.63966 | + | AVERAGEMASS 582.63966 |
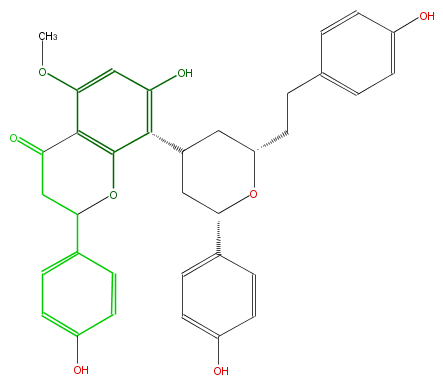
| − | SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC | + | SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-0.7383 -0.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0241 -0.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7383 -1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0232 -2.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6911 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6906 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4058 -2.0676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1200 -1.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1195 -0.8297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4048 -0.4176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4061 -2.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0227 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6415 -3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7748 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4893 -0.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2037 -0.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2037 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4893 0.7861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7748 0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7969 0.7161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0241 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6904 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6904 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0241 2.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7386 1.5914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7386 0.7664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3981 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1125 1.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8270 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8270 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1125 3.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3981 2.8250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4092 3.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3934 1.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0684 1.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7577 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7577 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4721 3.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1866 2.8027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1866 1.9777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4721 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7969 3.1550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3844 -0.4583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
7 11 2 0 0 0 0
4 12 1 0 0 0 0
12 13 1 0 0 0 0
9 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
2 21 1 6 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
23 27 1 6 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
32 27 1 0 0 0 0
30 33 1 0 0 0 0
25 34 1 6 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
1 43 1 0 0 0 0
S SKP 8
ID FL2FBANC0005
KNApSAcK_ID C00014289
NAME Calyxin K
CAS_RN 332877-78-8
FORMULA C35H34O8
EXACTMASS 582.225368064
AVERAGEMASS 582.63966
SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC
M END