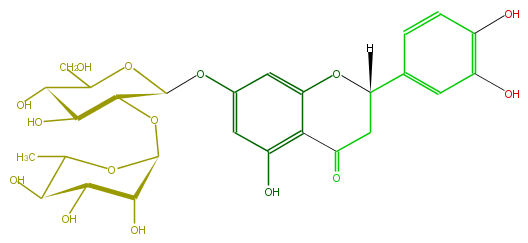
Mol:FL2FACGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4969 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4969 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4969 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4969 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1984 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1984 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8936 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8936 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8936 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8936 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1984 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1984 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5889 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5889 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2841 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2841 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2841 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2841 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5889 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5889 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9790 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9790 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6935 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6935 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4081 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4081 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4081 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4081 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6935 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6935 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9790 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9790 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2841 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2841 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1984 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1984 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1916 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1916 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9022 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9022 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3650 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3650 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7190 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7190 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9250 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9250 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6574 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6574 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4514 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4514 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1285 0.0290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1285 0.0290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8802 0.3362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8802 0.3362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5116 -0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5116 -0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5889 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5889 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9593 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9593 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4533 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4533 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5033 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5033 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5476 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5476 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0535 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0535 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0036 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0036 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5476 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5476 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9489 -1.9911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9489 -1.9911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0249 -1.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0249 -1.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6469 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6469 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1211 0.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1211 0.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1211 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1211 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0140 1.1176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0140 1.1176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1211 1.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1211 1.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 9 17 1 1 0 0 0 | + | 9 17 1 1 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 20 23 1 1 0 0 0 | + | 20 23 1 1 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 7 29 2 0 0 0 0 | + | 7 29 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 34 26 1 0 0 0 0 | + | 34 26 1 0 0 0 0 |
| − | 13 40 1 0 0 0 0 | + | 13 40 1 0 0 0 0 |
| − | 14 41 1 0 0 0 0 | + | 14 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 47 0.5626 -0.4284 | + | M SBV 1 47 0.5626 -0.4284 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FACGS0001 | + | ID FL2FACGS0001 |
| − | FORMULA C27H31HO15 | + | FORMULA C27H31HO15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES C(C5)(Oc(c(C5=O)4)cc(cc4O)OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)(c(c1)ccc(O)c1O)[H] | + | SMILES C(C5)(Oc(c(C5=O)4)cc(cc4O)OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)(c(c1)ccc(O)c1O)[H] |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.4969 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4969 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1984 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8936 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8936 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1984 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5889 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2841 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2841 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5889 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9790 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6935 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4081 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4081 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6935 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9790 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2841 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
0.1984 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1916 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9022 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3650 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7190 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9250 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6574 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4514 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1285 0.0290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8802 0.3362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5116 -0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5889 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9593 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4533 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5033 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5476 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0535 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0036 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5476 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9489 -1.9911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0249 -1.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6469 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1211 0.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1211 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0140 1.1176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1211 1.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
9 17 1 1 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
19 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
20 23 1 1 0 0 0
20 24 1 0 0 0 0
24 25 1 0 0 0 0
25 21 1 0 0 0 0
23 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
7 29 2 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
33 36 1 0 0 0 0
32 37 1 0 0 0 0
31 38 1 0 0 0 0
30 39 1 0 0 0 0
34 26 1 0 0 0 0
13 40 1 0 0 0 0
14 41 1 0 0 0 0
42 43 1 0 0 0 0
25 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^CH2OH
M SBV 1 47 0.5626 -0.4284
S SKP 5
ID FL2FACGS0001
FORMULA C27H31HO15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES C(C5)(Oc(c(C5=O)4)cc(cc4O)OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)(c(c1)ccc(O)c1O)[H]
M END