Mol:FL2FAAGC0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 46 0 0 0 0 0 0 0 0999 V2000 | + | 41 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5807 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5807 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5807 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5807 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0244 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0244 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4680 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4680 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4680 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4680 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0244 -0.8393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0244 -0.8393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0883 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0883 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6446 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6446 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6446 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6446 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0883 -0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0883 -0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2007 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2007 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7676 -1.1667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7676 -1.1667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3346 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3346 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3346 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3346 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7676 0.1426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7676 0.1426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2007 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2007 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0883 -2.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0883 -2.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9014 0.1425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9014 0.1425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1367 -0.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1367 -0.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0244 -2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0244 -2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2365 1.9127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2365 1.9127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6697 1.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6697 1.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3115 0.7095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3115 0.7095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4428 0.0770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4428 0.0770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8034 0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8034 0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2159 1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2159 1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4964 2.5017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4964 2.5017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9014 2.2207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9014 2.2207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3703 1.6239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3703 1.6239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7676 0.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7676 0.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3362 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3362 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3362 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3362 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7676 2.1103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7676 2.1103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1990 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1990 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1990 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1990 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7153 2.0613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7153 2.0613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7153 2.7185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7153 2.7185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1005 1.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1005 1.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1367 -2.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1367 -2.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7892 0.4337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7892 0.4337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7676 2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7676 2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 15 30 1 0 0 0 0 | + | 15 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 29 38 1 0 0 0 0 | + | 29 38 1 0 0 0 0 |
| − | 2 39 1 0 0 0 0 | + | 2 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FAAGC0001 | + | ID FL2FAAGC0001 |
| − | KNApSAcK_ID C00008388 | + | KNApSAcK_ID C00008388 |
| − | NAME Poriolide | + | NAME Poriolide |
| − | CAS_RN 39262-30-1 | + | CAS_RN 39262-30-1 |
| − | FORMULA C29H26O12 | + | FORMULA C29H26O12 |
| − | EXACTMASS 566.1424262959999 | + | EXACTMASS 566.1424262959999 |
| − | AVERAGEMASS 566.50954 | + | AVERAGEMASS 566.50954 |
| − | SMILES C(C62)C(c(c(O6)5)c(c(c(c5)OC(C(O)1)OC(COC(c(c4O)cc(cc4)c(c3)c(ccc23)O)=O)C(C(O)1)O)C)O)=O | + | SMILES C(C62)C(c(c(O6)5)c(c(c(c5)OC(C(O)1)OC(COC(c(c4O)cc(cc4)c(c3)c(ccc23)O)=O)C(C(O)1)O)C)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 46 0 0 0 0 0 0 0 0999 V2000
-1.5807 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5807 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0244 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4680 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4680 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0244 -0.8393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0883 -2.1240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6446 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6446 -1.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0883 -0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2007 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7676 -1.1667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3346 -0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3346 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7676 0.1426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2007 -0.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0883 -2.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9014 0.1425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1367 -0.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0244 -2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2365 1.9127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6697 1.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3115 0.7095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4428 0.0770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8034 0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2159 1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4964 2.5017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9014 2.2207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3703 1.6239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7676 0.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3362 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3362 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7676 2.1103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1990 1.7820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1990 1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7153 2.0613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7153 2.7185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1005 1.8427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1367 -2.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7892 0.4337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7676 2.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
26 29 1 0 0 0 0
19 24 1 0 0 0 0
15 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 30 1 0 0 0 0
34 36 1 0 0 0 0
36 37 2 0 0 0 0
36 38 1 0 0 0 0
29 38 1 0 0 0 0
2 39 1 0 0 0 0
23 40 1 0 0 0 0
33 41 1 0 0 0 0
S SKP 8
ID FL2FAAGC0001
KNApSAcK_ID C00008388
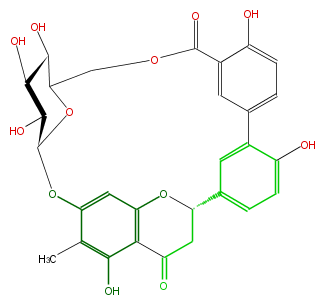
NAME Poriolide
CAS_RN 39262-30-1
FORMULA C29H26O12
EXACTMASS 566.1424262959999
AVERAGEMASS 566.50954
SMILES C(C62)C(c(c(O6)5)c(c(c(c5)OC(C(O)1)OC(COC(c(c4O)cc(cc4)c(c3)c(ccc23)O)=O)C(C(O)1)O)C)O)=O
M END