Mol:FL2FA9NC0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3682 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3682 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3682 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3682 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8856 -2.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8856 -2.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4030 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4030 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4030 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4030 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8856 -1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8856 -1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9208 -2.2233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9208 -2.2233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8856 -2.7804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8856 -2.7804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4397 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4397 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9573 -2.2241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9573 -2.2241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4748 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4748 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4748 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4748 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9573 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9573 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4397 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4397 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1265 -1.1874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1265 -1.1874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9573 -0.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9573 -0.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2687 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2687 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2522 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2522 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7730 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7730 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7730 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7730 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2522 1.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2522 1.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2687 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2687 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2522 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2522 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7891 1.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7891 1.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2522 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2522 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2457 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2457 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2457 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2457 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7652 2.7804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7652 2.7804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2847 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2847 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2847 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2847 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7652 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7652 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2090 2.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2090 2.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2939 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2939 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8148 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8148 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8148 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8148 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2939 1.0194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2939 1.0194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3352 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3352 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8517 0.7209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8517 0.7209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3682 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3682 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3682 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3682 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8517 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8517 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3352 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3352 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2939 -0.7074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2939 -0.7074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 3 8 1 0 0 0 0 | + | 3 8 1 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 12 15 1 0 0 0 0 | + | 12 15 1 0 0 0 0 |
| − | 13 16 1 0 0 0 0 | + | 13 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 19 33 1 0 0 0 0 | + | 19 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 20 1 0 0 0 0 | + | 36 20 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 33 43 2 0 0 0 0 | + | 33 43 2 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FA9NC0004 | + | ID FL2FA9NC0004 |
| − | KNApSAcK_ID C00008374 | + | KNApSAcK_ID C00008374 |
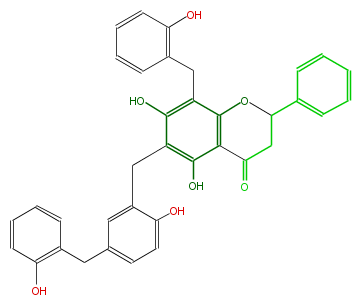
| − | NAME Uvarinol | + | NAME Uvarinol |
| − | CAS_RN 66754-55-0 | + | CAS_RN 66754-55-0 |
| − | FORMULA C36H30O7 | + | FORMULA C36H30O7 |
| − | EXACTMASS 574.199153314 | + | EXACTMASS 574.199153314 |
| − | AVERAGEMASS 574.6192 | + | AVERAGEMASS 574.6192 |
| − | SMILES C(c(c5)c(O)ccc(Cc(c6)c(O)ccc6)5)c(c(O)3)c(c(Cc(c4)c(ccc4)O)c(c13)OC(c(c2)cccc2)CC1=O)O | + | SMILES C(c(c5)c(O)ccc(Cc(c6)c(O)ccc6)5)c(c(O)3)c(c(Cc(c4)c(ccc4)O)c(c13)OC(c(c2)cccc2)CC1=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-3.3682 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3682 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8856 -2.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4030 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4030 -1.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8856 -1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9208 -2.2233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8856 -2.7804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4397 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9573 -2.2241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4748 -1.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4748 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9573 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4397 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1265 -1.1874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9573 -0.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2687 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2522 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7730 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7730 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2522 1.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2687 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2522 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7891 1.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2522 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2457 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2457 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7652 2.7804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2847 2.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2847 1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7652 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2090 2.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2939 -0.1835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8148 0.1172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8148 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2939 1.0194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3352 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8517 0.7209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3682 1.0191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3682 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8517 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3352 1.6156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2939 -0.7074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
3 8 1 0 0 0 0
7 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 9 1 0 0 0 0
12 15 1 0 0 0 0
13 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 21 1 0 0 0 0
21 22 2 0 0 0 0
22 17 1 0 0 0 0
18 23 1 0 0 0 0
22 24 1 0 0 0 0
21 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 26 1 0 0 0 0
27 32 1 0 0 0 0
19 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 20 1 0 0 0 0
35 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
33 43 2 0 0 0 0
S SKP 8
ID FL2FA9NC0004
KNApSAcK_ID C00008374
NAME Uvarinol
CAS_RN 66754-55-0
FORMULA C36H30O7
EXACTMASS 574.199153314
AVERAGEMASS 574.6192
SMILES C(c(c5)c(O)ccc(Cc(c6)c(O)ccc6)5)c(c(O)3)c(c(Cc(c4)c(ccc4)O)c(c13)OC(c(c2)cccc2)CC1=O)O
M END