Mol:FL2F9AGI0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2394 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2394 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5267 1.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5267 1.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2362 -0.0365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2362 -0.0365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5194 -0.4459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5194 -0.4459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8068 -0.0302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8068 -0.0302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8104 0.7948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8104 0.7948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0905 -0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0905 -0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6221 -0.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6221 -0.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6184 0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6184 0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0978 1.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0978 1.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2722 1.1820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2722 1.1820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9882 0.7722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9882 0.7722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7011 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7011 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6979 2.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6979 2.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2690 2.0069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2690 2.0069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0894 -1.0828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0894 -1.0828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5194 -1.1540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5194 -1.1540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9334 -0.4390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9334 -0.4390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2848 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2848 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9334 -1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9334 -1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7586 -1.7102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7586 -1.7102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7586 -2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7586 -2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3869 -1.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3869 -1.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5752 0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5752 0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5894 -0.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5894 -0.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1530 0.4626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1530 0.4626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9451 0.6941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9451 0.6941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9311 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9311 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3674 0.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3674 0.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3869 0.2672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3869 0.2672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1256 -0.2615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1256 -0.2615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0386 -0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0386 -0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1349 0.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1349 0.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3270 -1.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3270 -1.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1400 -1.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1400 -1.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4911 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4911 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1739 -0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1739 -0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3608 -0.4960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3608 -0.4960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0095 -1.2429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0095 -1.2429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3253 -1.1077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3253 -1.1077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6400 -2.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6400 -2.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0895 -2.2199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0895 -2.2199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 4 18 1 0 0 0 0 | + | 4 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 26 33 1 0 0 0 0 | + | 26 33 1 0 0 0 0 |
| − | 25 34 1 0 0 0 0 | + | 25 34 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2F9AGI0001 | + | ID FL2F9AGI0001 |
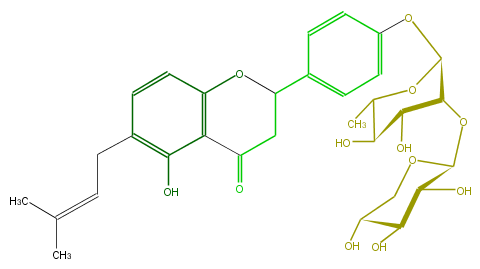
| − | FORMULA C31H38O12 | + | FORMULA C31H38O12 |
| − | EXACTMASS 602.23632668 | + | EXACTMASS 602.23632668 |
| − | AVERAGEMASS 602.62622 | + | AVERAGEMASS 602.62622 |
| − | SMILES C(=C(C)C)Cc(c1O)ccc(O2)c1C(CC2c(c5)ccc(c5)OC(O4)C(C(O)C(C4C)O)OC(O3)C(O)C(O)C(C3)O)=O | + | SMILES C(=C(C)C)Cc(c1O)ccc(O2)c1C(CC2c(c5)ccc(c5)OC(O4)C(C(O)C(C4C)O)OC(O3)C(O)C(O)C(C3)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.2394 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5267 1.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2362 -0.0365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5194 -0.4459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8068 -0.0302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8104 0.7948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0905 -0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6221 -0.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6184 0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0978 1.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2722 1.1820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9882 0.7722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7011 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6979 2.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2690 2.0069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0894 -1.0828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5194 -1.1540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9334 -0.4390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2848 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9334 -1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7586 -1.7102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7586 -2.4222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3869 -1.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5752 0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5894 -0.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1530 0.4626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9451 0.6941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9311 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3674 0.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3869 0.2672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1256 -0.2615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0386 -0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1349 0.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3270 -1.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1400 -1.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4911 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1739 -0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3608 -0.4960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0095 -1.2429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3253 -1.1077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6400 -2.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0895 -2.2199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
4 18 1 0 0 0 0
3 19 1 0 0 0 0
14 20 1 0 0 0 0
19 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
22 24 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 25 1 0 0 0 0
28 31 1 0 0 0 0
27 32 1 0 0 0 0
26 33 1 0 0 0 0
25 34 1 0 0 0 0
29 20 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
35 43 1 0 0 0 0
38 31 1 0 0 0 0
S SKP 5
ID FL2F9AGI0001
FORMULA C31H38O12
EXACTMASS 602.23632668
AVERAGEMASS 602.62622
SMILES C(=C(C)C)Cc(c1O)ccc(O2)c1C(CC2c(c5)ccc(c5)OC(O4)C(C(O)C(C4C)O)OC(O3)C(O)C(O)C(C3)O)=O
M END