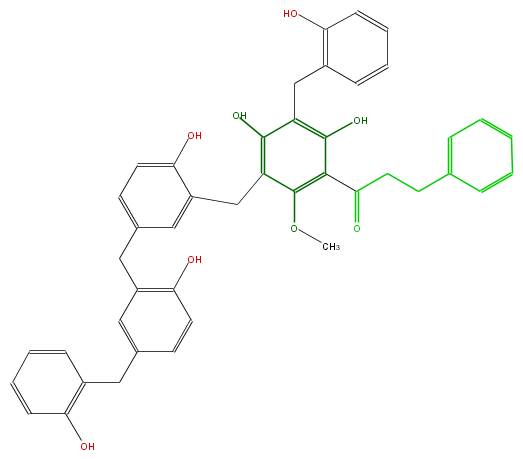
Mol:FL1DA9NC0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -6.3873 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3873 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3873 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3873 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6728 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6728 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9583 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9583 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9583 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9583 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6728 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6728 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2439 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2439 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5294 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5294 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5294 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5294 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8149 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8149 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1005 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1005 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1005 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1005 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8149 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8149 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6728 3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6728 3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8149 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8149 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1005 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1005 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1005 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1005 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3860 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3860 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6715 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6715 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6715 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6715 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3860 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3860 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3860 0.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3860 0.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0430 -1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0430 -1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1501 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1501 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0248 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0248 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2623 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2623 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6748 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6748 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2623 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2623 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4373 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4373 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1551 -1.7314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1551 -1.7314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6748 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6748 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4998 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4998 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4998 -0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4998 -0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2623 1.9017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2623 1.9017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4998 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4998 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9123 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9123 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1498 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1498 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3873 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3873 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9748 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9748 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1498 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1498 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9123 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9123 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7373 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7373 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1498 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1498 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9123 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9123 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4998 -3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4998 -3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0248 1.1872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0248 1.1872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 8 1 0 0 0 0 | + | 13 8 1 0 0 0 0 |
| − | 6 14 1 0 0 0 0 | + | 6 14 1 0 0 0 0 |
| − | 10 15 1 0 0 0 0 | + | 10 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 16 1 0 0 0 0 | + | 21 16 1 0 0 0 0 |
| − | 11 22 1 0 0 0 0 | + | 11 22 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 33 45 2 0 0 0 0 | + | 33 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 33 1 0 0 0 0 | + | 49 33 1 0 0 0 0 |
| − | 45 50 1 0 0 0 0 | + | 45 50 1 0 0 0 0 |
| − | 30 51 1 0 0 0 0 | + | 30 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1DA9NC0015 | + | ID FL1DA9NC0015 |
| − | KNApSAcK_ID C00014642 | + | KNApSAcK_ID C00014642 |
| − | NAME 2',4'-Dihydroxy-6'-methoxy-3'-(2-hydroxybenzyl)-5'-(3 x 2-hydroxybenzyl)dihydrochalcone | + | NAME 2',4'-Dihydroxy-6'-methoxy-3'-(2-hydroxybenzyl)-5'-(3 x 2-hydroxybenzyl)dihydrochalcone |
| − | CAS_RN 192222-56-3 | + | CAS_RN 192222-56-3 |
| − | FORMULA C44H40O8 | + | FORMULA C44H40O8 |
| − | EXACTMASS 696.272318256 | + | EXACTMASS 696.272318256 |
| − | AVERAGEMASS 696.7836 | + | AVERAGEMASS 696.7836 |
| − | SMILES COc(c3Cc(c4)c(ccc4Cc(c5)c(ccc5Cc(c6)c(O)ccc6)O)O)c(c(c(c(O)3)Cc(c2O)cccc2)O)C(=O)CCc(c1)cccc1 | + | SMILES COc(c3Cc(c4)c(ccc4Cc(c5)c(ccc5Cc(c6)c(O)ccc6)O)O)c(c(c(c(O)3)Cc(c2O)cccc2)O)C(=O)CCc(c1)cccc1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-6.3873 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.3873 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6728 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9583 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9583 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6728 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2439 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5294 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5294 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8149 0.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1005 1.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1005 1.8621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8149 2.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6728 3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8149 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1005 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1005 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3860 -1.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6715 -1.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6715 -0.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3860 -0.2004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3860 0.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0430 -1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1501 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0248 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2623 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6748 -0.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2623 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4373 0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1551 -1.7314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6748 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4998 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4998 -0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2623 1.9017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4998 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9123 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1498 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9748 1.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3873 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9748 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1498 2.6162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9123 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7373 -2.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1498 -1.6707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7373 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9123 -0.9562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4998 -3.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0248 1.1872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4373 1.9017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 8 1 0 0 0 0
6 14 1 0 0 0 0
10 15 1 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 16 1 0 0 0 0
11 22 1 0 0 0 0
19 23 1 0 0 0 0
20 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
26 31 1 0 0 0 0
27 32 1 0 0 0 0
32 33 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 39 1 0 0 0 0
33 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 33 1 0 0 0 0
45 50 1 0 0 0 0
30 51 1 0 0 0 0
51 52 1 0 0 0 0
S SKP 8
ID FL1DA9NC0015
KNApSAcK_ID C00014642
NAME 2',4'-Dihydroxy-6'-methoxy-3'-(2-hydroxybenzyl)-5'-(3 x 2-hydroxybenzyl)dihydrochalcone
CAS_RN 192222-56-3
FORMULA C44H40O8
EXACTMASS 696.272318256
AVERAGEMASS 696.7836
SMILES COc(c3Cc(c4)c(ccc4Cc(c5)c(ccc5Cc(c6)c(O)ccc6)O)O)c(c(c(c(O)3)Cc(c2O)cccc2)O)C(=O)CCc(c1)cccc1
M END