Mol:FL1CHYNS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 31 32 0 0 0 0 0 0 0 0999 V2000 | + | 31 32 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6887 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6887 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6887 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6887 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1324 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1324 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5761 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5761 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5761 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5761 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1324 0.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1324 0.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0198 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0198 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4635 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4635 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0926 -0.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0926 -0.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6487 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6487 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2097 -0.6160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2097 -0.6160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7707 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7707 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7707 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7707 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2097 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2097 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6487 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6487 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0198 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0198 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0926 -1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0926 -1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5689 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5689 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8544 -1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8544 -1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0460 0.9690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0460 0.9690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5460 1.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5460 1.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4901 1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4901 1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9288 2.1525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9288 2.1525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8544 1.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8544 1.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1399 0.8319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1399 0.8319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6368 0.8555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6368 0.8555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5028 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5028 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7756 -0.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7756 -0.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4901 -1.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4901 -1.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6368 -0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6368 -0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5028 -1.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5028 -1.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 9 17 2 0 0 0 0 | + | 9 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 6 24 1 0 0 0 0 | + | 6 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 13 26 1 0 0 0 0 | + | 13 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 11 28 1 0 0 0 0 | + | 11 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 12 30 1 0 0 0 0 | + | 12 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | M STY 1 7 SUP | + | M STY 1 7 SUP |
| − | M SLB 1 7 7 | + | M SLB 1 7 7 |
| − | M SAL 7 2 30 31 | + | M SAL 7 2 30 31 |
| − | M SBL 7 1 31 | + | M SBL 7 1 31 |
| − | M SMT 7 OCH3 | + | M SMT 7 OCH3 |
| − | M SVB 7 31 1.9743 -0.4984 | + | M SVB 7 31 1.9743 -0.4984 |
| − | M STY 1 6 SUP | + | M STY 1 6 SUP |
| − | M SLB 1 6 6 | + | M SLB 1 6 6 |
| − | M SAL 6 2 28 29 | + | M SAL 6 2 28 29 |
| − | M SBL 6 1 29 | + | M SBL 6 1 29 |
| − | M SMT 6 OCH3 | + | M SMT 6 OCH3 |
| − | M SVB 6 29 0.7756 -0.7779 | + | M SVB 6 29 0.7756 -0.7779 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 26 27 | + | M SAL 5 2 26 27 |
| − | M SBL 5 1 27 | + | M SBL 5 1 27 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 27 2.3315 0.6795 | + | M SVB 5 27 2.3315 0.6795 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 24 25 | + | M SAL 4 2 24 25 |
| − | M SBL 4 1 25 | + | M SBL 4 1 25 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 25 -1.8544 1.2444 | + | M SVB 4 25 -1.8544 1.2444 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 22 23 | + | M SAL 3 2 22 23 |
| − | M SBL 3 1 23 | + | M SBL 3 1 23 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 23 1.4901 1.2539 | + | M SVB 3 23 1.4901 1.2539 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 20 21 | + | M SAL 2 2 20 21 |
| − | M SBL 2 1 21 | + | M SBL 2 1 21 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 21 -3.046 0.969 | + | M SVB 2 21 -3.046 0.969 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 18 19 | + | M SAL 1 2 18 19 |
| − | M SBL 1 1 19 | + | M SBL 1 1 19 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 19 -2.5689 -0.7738 | + | M SVB 1 19 -2.5689 -0.7738 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CHYNS0006 | + | ID FL1CHYNS0006 |
| − | KNApSAcK_ID C00006998 | + | KNApSAcK_ID C00006998 |
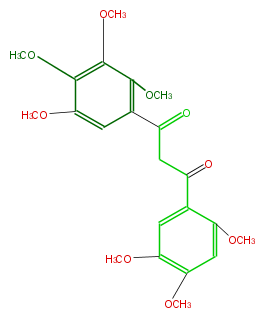
| − | NAME beta-Hydroxy-2,3,4,5,2',4',5'-heptamethoxychalcone | + | NAME beta-Hydroxy-2,3,4,5,2',4',5'-heptamethoxychalcone |
| − | CAS_RN 112470-94-7 | + | CAS_RN 112470-94-7 |
| − | FORMULA C22H26O9 | + | FORMULA C22H26O9 |
| − | EXACTMASS 434.15768243 | + | EXACTMASS 434.15768243 |
| − | AVERAGEMASS 434.43644 | + | AVERAGEMASS 434.43644 |
| − | SMILES COc(c(OC)1)c(OC)c(cc1C(=O)CC(=O)c(c(OC)2)cc(c(OC)c2)OC)OC | + | SMILES COc(c(OC)1)c(OC)c(cc1C(=O)CC(=O)c(c(OC)2)cc(c(OC)c2)OC)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
31 32 0 0 0 0 0 0 0 0999 V2000
-2.6887 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6887 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1324 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5761 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5761 0.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1324 0.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0198 -0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4635 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0926 -0.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6487 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2097 -0.6160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7707 -0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7707 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2097 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6487 0.3556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0198 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0926 -1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5689 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8544 -1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0460 0.9690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5460 1.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4901 1.2539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9288 2.1525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8544 1.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1399 0.8319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6368 0.8555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5028 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7756 -0.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4901 -1.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6368 -0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5028 -1.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
9 17 2 0 0 0 0
3 18 1 0 0 0 0
18 19 1 0 0 0 0
1 20 1 0 0 0 0
20 21 1 0 0 0 0
14 22 1 0 0 0 0
22 23 1 0 0 0 0
6 24 1 0 0 0 0
24 25 1 0 0 0 0
13 26 1 0 0 0 0
26 27 1 0 0 0 0
11 28 1 0 0 0 0
28 29 1 0 0 0 0
12 30 1 0 0 0 0
30 31 1 0 0 0 0
M STY 1 7 SUP
M SLB 1 7 7
M SAL 7 2 30 31
M SBL 7 1 31
M SMT 7 OCH3
M SVB 7 31 1.9743 -0.4984
M STY 1 6 SUP
M SLB 1 6 6
M SAL 6 2 28 29
M SBL 6 1 29
M SMT 6 OCH3
M SVB 6 29 0.7756 -0.7779
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 26 27
M SBL 5 1 27
M SMT 5 OCH3
M SVB 5 27 2.3315 0.6795
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 24 25
M SBL 4 1 25
M SMT 4 OCH3
M SVB 4 25 -1.8544 1.2444
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 22 23
M SBL 3 1 23
M SMT 3 OCH3
M SVB 3 23 1.4901 1.2539
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 20 21
M SBL 2 1 21
M SMT 2 OCH3
M SVB 2 21 -3.046 0.969
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 18 19
M SBL 1 1 19
M SMT 1 OCH3
M SVB 1 19 -2.5689 -0.7738
S SKP 8
ID FL1CHYNS0006
KNApSAcK_ID C00006998
NAME beta-Hydroxy-2,3,4,5,2',4',5'-heptamethoxychalcone
CAS_RN 112470-94-7
FORMULA C22H26O9
EXACTMASS 434.15768243
AVERAGEMASS 434.43644
SMILES COc(c(OC)1)c(OC)c(cc1C(=O)CC(=O)c(c(OC)2)cc(c(OC)c2)OC)OC
M END