Mol:FL1C3CGS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 47 0 0 0 0 0 0 0 0999 V2000 | + | 44 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.9924 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9924 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9924 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9924 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7069 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7069 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4213 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4213 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4213 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4213 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7069 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7069 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1358 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1358 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8503 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8503 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8503 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8503 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5647 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5647 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2792 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2792 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9937 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9937 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9937 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9937 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2792 1.2819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2792 1.2819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5647 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5647 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1358 -2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1358 -2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3800 0.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3800 0.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9020 -0.4623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9020 -0.4623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1268 -0.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1268 -0.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3155 -0.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3155 -0.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7935 0.3426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7935 0.3426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5687 0.0593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5687 0.0593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7801 0.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7801 0.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8156 -0.3162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8156 -0.3162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3730 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3730 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2373 -0.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2373 -0.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7069 -2.3169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7069 -2.3169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3244 0.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3244 0.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6749 1.2627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6749 1.2627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2549 -1.5231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2549 -1.5231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2792 2.0079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2792 2.0079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4132 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4132 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8251 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8251 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4132 2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4132 2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6490 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6490 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0610 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0610 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8849 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8849 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2974 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2974 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1224 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1224 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5349 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5349 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1224 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1224 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2974 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2974 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.3587 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.3587 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3587 0.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3587 0.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 17 24 1 0 0 0 0 | + | 17 24 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 3 27 1 0 0 0 0 | + | 3 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 32 23 1 0 0 0 0 | + | 32 23 1 0 0 0 0 |
| − | 29 44 1 0 0 0 0 | + | 29 44 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3CGS0024 | + | ID FL1C3CGS0024 |
| − | KNApSAcK_ID C00014512 | + | KNApSAcK_ID C00014512 |
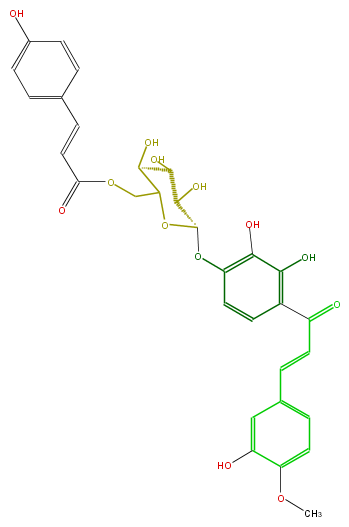
| − | NAME Okanin 4-methyl ether 4'-O-(6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxy-4-methoxychalcone 4'-O-(6''-O-p-coumaroylglucoside) | + | NAME Okanin 4-methyl ether 4'-O-(6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxy-4-methoxychalcone 4'-O-(6''-O-p-coumaroylglucoside) |
| − | CAS_RN 142628-32-8 | + | CAS_RN 142628-32-8 |
| − | FORMULA C31H30O13 | + | FORMULA C31H30O13 |
| − | EXACTMASS 610.168641046 | + | EXACTMASS 610.168641046 |
| − | AVERAGEMASS 610.5621 | + | AVERAGEMASS 610.5621 |
| − | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(c(c2)C(C=Cc(c1)cc(c(c1)OC)O)=O)O)O | + | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(c(c2)C(C=Cc(c1)cc(c(c1)OC)O)=O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 47 0 0 0 0 0 0 0 0999 V2000
0.9924 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9924 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7069 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4213 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4213 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7069 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1358 -1.6056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8503 -1.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8503 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5647 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2792 -0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9937 0.0444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9937 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2792 1.2819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5647 0.8694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1358 -2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3800 0.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9020 -0.4623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1268 -0.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3155 -0.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7935 0.3426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5687 0.0593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7801 0.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8156 -0.3162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3730 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2373 -0.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7069 -2.3169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3244 0.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6749 1.2627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2549 -1.5231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2792 2.0079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4132 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8251 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4132 2.4306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6490 1.7171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0610 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8849 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2974 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1224 1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5349 1.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1224 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2974 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.3587 1.0036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.3587 0.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 18 1 1 0 0 0
18 19 1 1 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 17 1 0 0 0 0
22 23 1 0 0 0 0
17 24 1 0 0 0 0
18 25 1 0 0 0 0
19 26 1 0 0 0 0
3 27 1 0 0 0 0
1 28 1 0 0 0 0
13 29 1 0 0 0 0
2 30 1 0 0 0 0
14 31 1 0 0 0 0
20 28 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
32 23 1 0 0 0 0
29 44 1 0 0 0 0
S SKP 8
ID FL1C3CGS0024
KNApSAcK_ID C00014512
NAME Okanin 4-methyl ether 4'-O-(6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxy-4-methoxychalcone 4'-O-(6''-O-p-coumaroylglucoside)
CAS_RN 142628-32-8
FORMULA C31H30O13
EXACTMASS 610.168641046
AVERAGEMASS 610.5621
SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(c(c2)C(C=Cc(c1)cc(c(c1)OC)O)=O)O)O
M END