Mol:BMMCTZ--e006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 34 0 0 1 0 0 0 0 0999 V2000 | + | 33 34 0 0 1 0 0 0 0 0999 V2000 |
| − | 3.7321 0.8945 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 0.8945 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -0.6055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -0.6055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 -0.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 -0.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 0.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 0.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -2.1055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -2.1055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9128 -0.2691 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9128 -0.2691 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2437 -1.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2437 -1.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 -0.6055 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 -0.6055 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4347 0.3890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4347 0.3890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4128 0.5969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4128 0.5969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6915 1.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6915 1.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8195 1.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8195 1.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8141 1.6150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8141 1.6150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5616 -3.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5616 -3.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6106 -3.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6106 -3.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4026 -2.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4026 -2.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4516 -1.9904 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 7.4516 -1.9904 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 9.2208 2.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2208 2.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2153 2.6331 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2153 2.6331 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3198 1.6386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3198 1.6386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1108 3.6276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1108 3.6276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2098 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2098 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6166 3.6512 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6166 3.6512 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5301 3.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5301 3.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7030 4.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7030 4.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.0233 4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.0233 4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7695 -4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7695 -4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3048 -2.9174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3048 -2.9174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7084 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7084 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12 11 2 0 0 0 0 | + | 12 11 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 12 9 1 0 0 0 0 | + | 12 9 1 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 11 21 1 0 0 0 0 | + | 11 21 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 13 15 1 0 0 0 0 | + | 13 15 1 0 0 0 0 |
| − | 9 5 1 0 0 0 0 | + | 9 5 1 0 0 0 0 |
| − | 21 20 1 0 0 0 0 | + | 21 20 1 0 0 0 0 |
| − | 21 33 1 4 0 0 0 | + | 21 33 1 4 0 0 0 |
| − | 14 16 1 0 0 0 0 | + | 14 16 1 0 0 0 0 |
| − | 5 4 1 0 0 0 0 | + | 5 4 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 4 3 2 0 0 0 0 | + | 4 3 2 0 0 0 0 |
| − | 4 8 1 0 0 0 0 | + | 4 8 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 17 22 1 0 0 0 0 | + | 17 22 1 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 18 32 1 0 0 0 0 | + | 18 32 1 0 0 0 0 |
| − | 18 31 2 0 0 0 0 | + | 18 31 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 2 0 0 0 0 | + | 23 25 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 27 30 2 0 0 0 0 | + | 27 30 2 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMMCTZ--e006 | + | ID BMMCTZ--e006 |
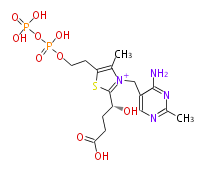
| − | NAME 2-(Succinyl)-thiamine diphosphate | + | NAME 2-(Succinyl)-thiamine diphosphate |
| − | FORMULA C16H25N4O10P2S | + | FORMULA C16H25N4O10P2S |
| − | EXACTMASS 527.0766 | + | EXACTMASS 527.0766 |
| − | AVERAGEMASS 527.4041 | + | AVERAGEMASS 527.4041 |
| − | SMILES OC(=O)CCC(O)c(s1)[n+1](Cc(c2)c(N)nc(C)n2)c(C)c(CCOP(O)(=O)OP(O)(O)=O)1 | + | SMILES OC(=O)CCC(O)c(s1)[n+1](Cc(c2)c(N)nc(C)n2)c(C)c(CCOP(O)(=O)OP(O)(O)=O)1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05381 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05381 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 34 0 0 1 0 0 0 0 0999 V2000
3.7321 0.8945 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -0.6055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 -0.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 0.3945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 0.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -2.1055 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9128 -0.2691 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
7.2437 -1.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 -0.6055 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0
6.4347 0.3890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4128 0.5969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6915 1.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8195 1.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8141 1.6150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.5616 -3.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.6106 -3.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4026 -2.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4516 -1.9904 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
9.2208 2.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.2153 2.6331 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
10.3198 1.6386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.1108 3.6276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.2098 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.6166 3.6512 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
12.5301 3.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.7030 4.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.0233 4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.7695 -4.5647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.3048 -2.9174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.7084 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12 11 2 0 0 0 0
12 13 1 0 0 0 0
12 9 1 0 0 0 0
11 10 1 0 0 0 0
11 21 1 0 0 0 0
13 14 2 0 0 0 0
13 15 1 0 0 0 0
9 5 1 0 0 0 0
21 20 1 0 0 0 0
21 33 1 4 0 0 0
14 16 1 0 0 0 0
5 4 1 0 0 0 0
5 6 2 0 0 0 0
20 19 1 0 0 0 0
16 17 1 0 0 0 0
4 3 2 0 0 0 0
4 8 1 0 0 0 0
6 1 1 0 0 0 0
19 18 1 0 0 0 0
17 22 1 0 0 0 0
3 2 1 0 0 0 0
18 32 1 0 0 0 0
18 31 2 0 0 0 0
22 23 1 0 0 0 0
2 7 1 0 0 0 0
23 26 1 0 0 0 0
23 24 1 0 0 0 0
23 25 2 0 0 0 0
26 27 1 0 0 0 0
27 29 1 0 0 0 0
27 28 1 0 0 0 0
27 30 2 0 0 0 0
10 14 1 0 0 0 0
1 2 2 0 0 0 0
S SKP 7
ID BMMCTZ--e006
NAME 2-(Succinyl)-thiamine diphosphate
FORMULA C16H25N4O10P2S
EXACTMASS 527.0766
AVERAGEMASS 527.4041
SMILES OC(=O)CCC(O)c(s1)[n+1](Cc(c2)c(N)nc(C)n2)c(C)c(CCOP(O)(=O)OP(O)(O)=O)1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C05381
M END