Mol:BMMCPYURS601
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 68 70 0 0 1 0 0 0 0 0999 V2000 | + | 68 70 0 0 1 0 0 0 0 0999 V2000 |
| − | 4.9836 -3.1164 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.9836 -3.1164 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 5.3903 -2.2028 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.3903 -2.2028 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.8025 -1.3938 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.8025 -1.3938 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.8080 -1.4983 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8080 -1.4983 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.4013 -2.4119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.4013 -2.4119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4067 -2.5164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4067 -2.5164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5714 -3.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5714 -3.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3848 -2.0983 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3848 -2.0983 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7916 -1.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7916 -1.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7861 -1.0802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7861 -1.0802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1928 -0.1667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 8.1928 -0.1667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 9.1873 -0.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1873 -0.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5941 0.8514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5941 0.8514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5886 0.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5886 0.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9953 1.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9953 1.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9899 1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9899 1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3966 2.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.3966 2.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3911 2.9921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3911 2.9921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.7979 3.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.7979 3.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7924 4.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7924 4.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.1991 4.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1991 4.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1936 5.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1936 5.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2038 -0.3757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2038 -0.3757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6050 0.6423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6050 0.6423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6215 0.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6215 0.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0282 1.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0282 1.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4404 2.0513 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.4404 2.0513 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.8472 2.9649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8472 2.9649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2594 3.7739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2594 3.7739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6661 4.6874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6661 4.6874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0783 5.4965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0783 5.4965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4851 6.4100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4851 6.4100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8973 7.2190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8973 7.2190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3040 8.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3040 8.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7162 8.9416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7162 8.9416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1230 9.8551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1230 9.8551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5352 10.6641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5352 10.6641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9419 11.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9419 11.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6269 0.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6269 0.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2093 -0.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2093 -0.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4459 1.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4459 1.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2202 -0.6893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2202 -0.6893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9890 -3.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9890 -3.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8183 -8.5777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8183 -8.5777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9522 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9522 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9522 -10.0777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9522 -10.0777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8183 -10.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8183 -10.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6843 -10.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6843 -10.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6843 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6843 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0862 -8.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0862 -8.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8183 -11.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8183 -11.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8183 -7.5777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 10.8183 -7.5777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 11.6273 -6.9899 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 11.6273 -6.9899 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 11.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 11.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 10.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 10.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 9.7305 -5.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7305 -5.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6273 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.6273 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9061 -5.2298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9061 -5.2298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0093 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0093 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7360 -5.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7360 -5.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1482 -4.5253 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1482 -4.5253 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3392 -5.1131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3392 -5.1131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9572 -3.9376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9572 -3.9376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5604 -3.7163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5604 -3.7163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5659 -3.8208 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5659 -3.8208 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6704 -4.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6704 -4.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4613 -2.8263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4613 -2.8263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 25 39 2 0 0 0 0 | + | 25 39 2 0 0 0 0 |
| − | 26 25 1 0 0 0 0 | + | 26 25 1 0 0 0 0 |
| − | 27 41 1 4 0 0 0 | + | 27 41 1 4 0 0 0 |
| − | 5 43 1 6 0 0 0 | + | 5 43 1 6 0 0 0 |
| − | 5 4 1 0 0 0 0 | + | 5 4 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 1 43 1 0 0 0 0 | + | 1 43 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 1 7 1 4 0 0 0 | + | 1 7 1 4 0 0 0 |
| − | 3 40 1 6 0 0 0 | + | 3 40 1 6 0 0 0 |
| − | 4 42 1 1 0 0 0 | + | 4 42 1 1 0 0 0 |
| − | 2 8 1 1 0 0 0 | + | 2 8 1 1 0 0 0 |
| − | 6 44 1 0 0 0 0 | + | 6 44 1 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 9 23 2 0 0 0 0 | + | 9 23 2 0 0 0 0 |
| − | 10 9 1 0 0 0 0 | + | 10 9 1 0 0 0 0 |
| − | 11 24 1 4 0 0 0 | + | 11 24 1 4 0 0 0 |
| − | 56 60 1 6 0 0 0 | + | 56 60 1 6 0 0 0 |
| − | 56 55 1 0 0 0 0 | + | 56 55 1 0 0 0 0 |
| − | 53 60 1 6 0 0 0 | + | 53 60 1 6 0 0 0 |
| − | 55 54 1 0 0 0 0 | + | 55 54 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 53 45 1 0 0 0 0 | + | 53 45 1 0 0 0 0 |
| − | 54 58 1 1 0 0 0 | + | 54 58 1 1 0 0 0 |
| − | 55 59 1 1 0 0 0 | + | 55 59 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 47 46 1 0 0 0 0 | + | 47 46 1 0 0 0 0 |
| − | 46 45 1 0 0 0 0 | + | 46 45 1 0 0 0 0 |
| − | 45 50 1 0 0 0 0 | + | 45 50 1 0 0 0 0 |
| − | 50 49 2 0 0 0 0 | + | 50 49 2 0 0 0 0 |
| − | 49 48 1 0 0 0 0 | + | 49 48 1 0 0 0 0 |
| − | 48 52 2 0 0 0 0 | + | 48 52 2 0 0 0 0 |
| − | 46 51 2 0 0 0 0 | + | 46 51 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 61 1 0 0 0 0 | + | 57 61 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 65 1 0 0 0 0 | + | 62 65 1 0 0 0 0 |
| − | 66 65 1 0 0 0 0 | + | 66 65 1 0 0 0 0 |
| − | 62 64 1 0 0 0 0 | + | 62 64 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 66 7 1 0 0 0 0 | + | 66 7 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 66 68 2 0 0 0 0 | + | 66 68 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMMCPYURS601 | + | ID BMMCPYURS601 |
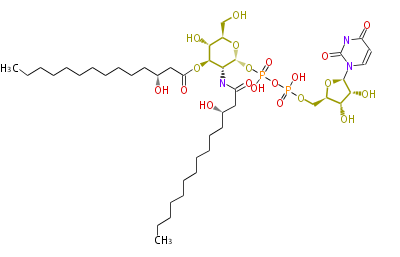
| − | NAME UDP-2,3-bis(3-hydroxy-tetradecanoyl)-glucosamine | + | NAME UDP-2,3-bis(3-hydroxy-tetradecanoyl)-glucosamine |
| − | FORMULA C43H77N3O20P2 | + | FORMULA C43H77N3O20P2 |
| − | EXACTMASS 1017.4575 | + | EXACTMASS 1017.4575 |
| − | AVERAGEMASS 1018.0272 | + | AVERAGEMASS 1018.0272 |
| − | SMILES O=C(CC(CCCCCCCCCCC)O)O[C@@H]([C@@H]1O)[C@H](C(OP(O)(=O)OP(OC[C@H]([C@@H](O)2)O[C@@H](N(C=3)C(=O)NC(C3)=O)[C@@H]2O)(O)=O)O[C@H](CO)1)NC(=O)CC(O)CCCCCCCCCCC | + | SMILES O=C(CC(CCCCCCCCCCC)O)O[C@@H]([C@@H]1O)[C@H](C(OP(O)(=O)OP(OC[C@H]([C@@H](O)2)O[C@@H](N(C=3)C(=O)NC(C3)=O)[C@@H]2O)(O)=O)O[C@H](CO)1)NC(=O)CC(O)CCCCCCCCCCC |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04652 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04652 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
68 70 0 0 1 0 0 0 0 0999 V2000
4.9836 -3.1164 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
5.3903 -2.2028 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.8025 -1.3938 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.8080 -1.4983 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.4013 -2.4119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4067 -2.5164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5714 -3.9254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3848 -2.0983 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.7916 -1.1847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7861 -1.0802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1928 -0.1667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
9.1873 -0.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.5941 0.8514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5886 0.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.9953 1.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.9899 1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.3966 2.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3911 2.9921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.7979 3.9056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.7924 4.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.1991 4.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.1936 5.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2038 -0.3757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.6050 0.6423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6215 0.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0282 1.2423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4404 2.0513 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.8472 2.9649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2594 3.7739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6661 4.6874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0783 5.4965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4851 6.4100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8973 7.2190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3040 8.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7162 8.9416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1230 9.8551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5352 10.6641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9419 11.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6269 0.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2093 -0.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4459 1.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2202 -0.6893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9890 -3.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.8183 -8.5777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.9522 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9522 -10.0777 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.8183 -10.5777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6843 -10.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6843 -9.0777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0862 -8.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.8183 -11.5777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.8183 -7.5777 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
11.6273 -6.9899 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
11.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
10.3183 -6.0388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
9.7305 -5.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.6273 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.9061 -5.2298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.0093 -6.9899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.7360 -5.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.1482 -4.5253 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
7.3392 -5.1131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.9572 -3.9376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.5604 -3.7163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.5659 -3.8208 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
6.6704 -4.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.4613 -2.8263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
25 40 1 0 0 0 0
25 39 2 0 0 0 0
26 25 1 0 0 0 0
27 41 1 4 0 0 0
5 43 1 6 0 0 0
5 4 1 0 0 0 0
4 3 1 0 0 0 0
3 2 1 0 0 0 0
2 1 1 0 0 0 0
1 43 1 0 0 0 0
5 6 1 0 0 0 0
1 7 1 4 0 0 0
3 40 1 6 0 0 0
4 42 1 1 0 0 0
2 8 1 1 0 0 0
6 44 1 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 14 1 0 0 0 0
14 15 1 0 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
9 8 1 0 0 0 0
9 23 2 0 0 0 0
10 9 1 0 0 0 0
11 24 1 4 0 0 0
56 60 1 6 0 0 0
56 55 1 0 0 0 0
53 60 1 6 0 0 0
55 54 1 0 0 0 0
53 54 1 0 0 0 0
53 45 1 0 0 0 0
54 58 1 1 0 0 0
55 59 1 1 0 0 0
47 48 1 0 0 0 0
47 46 1 0 0 0 0
46 45 1 0 0 0 0
45 50 1 0 0 0 0
50 49 2 0 0 0 0
49 48 1 0 0 0 0
48 52 2 0 0 0 0
46 51 2 0 0 0 0
56 57 1 0 0 0 0
57 61 1 0 0 0 0
61 62 1 0 0 0 0
62 65 1 0 0 0 0
66 65 1 0 0 0 0
62 64 1 0 0 0 0
62 63 2 0 0 0 0
66 7 1 0 0 0 0
66 67 1 0 0 0 0
66 68 2 0 0 0 0
S SKP 7
ID BMMCPYURS601
NAME UDP-2,3-bis(3-hydroxy-tetradecanoyl)-glucosamine
FORMULA C43H77N3O20P2
EXACTMASS 1017.4575
AVERAGEMASS 1018.0272
SMILES O=C(CC(CCCCCCCCCCC)O)O[C@@H]([C@@H]1O)[C@H](C(OP(O)(=O)OP(OC[C@H]([C@@H](O)2)O[C@@H](N(C=3)C(=O)NC(C3)=O)[C@@H]2O)(O)=O)O[C@H](CO)1)NC(=O)CC(O)CCCCCCCCCCC
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04652
M END