Mol:BMCCPPBL0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 46 0 0 0 0 0 0 0 0999 V2000 | + | 43 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.7246 -2.2374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7246 -2.2374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4156 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4156 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2246 -3.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2246 -3.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0337 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0337 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9847 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9847 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7279 -2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7279 -2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7060 -3.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7060 -3.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2060 -2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2060 -2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5369 -1.4271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5369 -1.4271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7448 -0.4489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7448 -0.4489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0016 0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0016 0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1062 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1062 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1926 1.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1926 1.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5235 0.8783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5235 0.8783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3554 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3554 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9487 2.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9487 2.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4487 2.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4487 2.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7796 3.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7796 3.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 3.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 3.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7246 -2.2374 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7246 -2.2374 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6233 -1.8338 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6233 -1.8338 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0235 0.0123 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0235 0.0123 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9706 2.2277 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9706 2.2277 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4646 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4646 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2246 -4.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2246 -4.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0907 -5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0907 -5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1127 -3.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1127 -3.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2005 -2.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2005 -2.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6073 -1.1521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6073 -1.1521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6018 -1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6018 -1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9722 1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9722 1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9722 2.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9722 2.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8382 3.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8382 3.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9847 2.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9847 2.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4432 2.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4432 2.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9875 4.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9875 4.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2443 5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2443 5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1369 -1.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1369 -1.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1896 -1.8566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1896 -1.8566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.0085 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.0085 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7042 2.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7042 2.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8382 4.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8382 4.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 3 2 2 0 0 0 0 | + | 3 2 2 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 20 4 1 0 0 0 0 | + | 20 4 1 0 0 0 0 |
| − | 6 5 1 0 0 0 0 | + | 6 5 1 0 0 0 0 |
| − | 5 4 2 0 0 0 0 | + | 5 4 2 0 0 0 0 |
| − | 6 21 2 0 0 0 0 | + | 6 21 2 0 0 0 0 |
| − | 21 9 1 0 0 0 0 | + | 21 9 1 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 8 7 2 0 0 0 0 | + | 8 7 2 0 0 0 0 |
| − | 7 6 1 0 0 0 0 | + | 7 6 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 23 16 1 0 0 0 0 | + | 23 16 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 22 11 1 0 0 0 0 | + | 22 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 10 9 2 0 0 0 0 | + | 10 9 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 19 43 2 0 0 0 0 | + | 19 43 2 0 0 0 0 |
| − | 1 38 2 0 0 0 0 | + | 1 38 2 0 0 0 0 |
| − | 12 31 1 0 0 0 0 | + | 12 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 8 28 1 0 0 0 0 | + | 8 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 30 40 2 0 0 0 0 | + | 30 40 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 33 42 2 0 0 0 0 | + | 33 42 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 13 34 1 0 0 0 0 | + | 13 34 1 0 0 0 0 |
| − | 17 35 1 0 0 0 0 | + | 17 35 1 0 0 0 0 |
| − | 18 36 1 0 0 0 0 | + | 18 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 2 24 1 0 0 0 0 | + | 2 24 1 0 0 0 0 |
| − | 3 25 1 0 0 0 0 | + | 3 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 7 27 1 0 0 0 0 | + | 7 27 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPBL0002 | + | ID BMCCPPBL0002 |
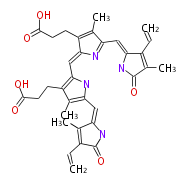
| − | NAME Biliverdin | + | NAME Biliverdin |
| − | FORMULA C33H34N4O6 | + | FORMULA C33H34N4O6 |
| − | EXACTMASS 582.2478 | + | EXACTMASS 582.2478 |
| − | AVERAGEMASS 582.6464 | + | AVERAGEMASS 582.6464 |
| − | SMILES n(c1=O)c(=Cc(n2)c(c(c2C=C(C=4CCC(O)=O)N=C(C4C)C=c(c(C=C)3)nc(=O)c(C)3)CCC(O)=O)C)c(c1C=C)C | + | SMILES n(c1=O)c(=Cc(n2)c(c(c2C=C(C=4CCC(O)=O)N=C(C4C)C=c(c(C=C)3)nc(=O)c(C)3)CCC(O)=O)C)c(c1C=C)C |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00500 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00500 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 46 0 0 0 0 0 0 0 0999 V2000
4.7246 -2.2374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4156 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2246 -3.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0337 -3.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9847 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7279 -2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7060 -3.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2060 -2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5369 -1.4271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7448 -0.4489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0016 0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1062 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1926 1.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5235 0.8783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3554 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9487 2.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4487 2.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7796 3.6289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 3.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7246 -2.2374 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.6233 -1.8338 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.0235 0.0123 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.9706 2.2277 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.4646 -3.4974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2246 -4.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0907 -5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1127 -3.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.2005 -2.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.6073 -1.1521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6018 -1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9722 1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9722 2.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.8382 3.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9847 2.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4432 2.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9875 4.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2443 5.2762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1369 -1.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.1896 -1.8566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.0085 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.7042 2.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.8382 4.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4 3 1 0 0 0 0
3 2 2 0 0 0 0
2 1 1 0 0 0 0
1 20 1 0 0 0 0
20 4 1 0 0 0 0
6 5 1 0 0 0 0
5 4 2 0 0 0 0
6 21 2 0 0 0 0
21 9 1 0 0 0 0
9 8 1 0 0 0 0
8 7 2 0 0 0 0
7 6 1 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 23 1 0 0 0 0
23 16 1 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
14 22 1 0 0 0 0
22 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
10 9 2 0 0 0 0
10 11 1 0 0 0 0
19 43 2 0 0 0 0
1 38 2 0 0 0 0
12 31 1 0 0 0 0
31 32 1 0 0 0 0
8 28 1 0 0 0 0
28 29 1 0 0 0 0
30 39 1 0 0 0 0
30 40 2 0 0 0 0
29 30 1 0 0 0 0
33 41 1 0 0 0 0
33 42 2 0 0 0 0
32 33 1 0 0 0 0
13 34 1 0 0 0 0
17 35 1 0 0 0 0
18 36 1 0 0 0 0
36 37 2 0 0 0 0
2 24 1 0 0 0 0
3 25 1 0 0 0 0
25 26 2 0 0 0 0
7 27 1 0 0 0 0
S SKP 7
ID BMCCPPBL0002
NAME Biliverdin
FORMULA C33H34N4O6
EXACTMASS 582.2478
AVERAGEMASS 582.6464
SMILES n(c1=O)c(=Cc(n2)c(c(c2C=C(C=4CCC(O)=O)N=C(C4C)C=c(c(C=C)3)nc(=O)c(C)3)CCC(O)=O)C)c(c1C=C)C
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00500
M END