Mol:BMCCCC--f026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 40 0 0 0 0 0 0 0 0999 V2000 | + | 36 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3917 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3917 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3917 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3917 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2579 -4.2208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2579 -4.2208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9157 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9157 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9157 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9157 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2579 -2.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2579 -2.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5642 -2.7024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5642 -2.7024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2162 -3.0755 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2162 -3.0755 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8502 -2.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8502 -2.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5629 -1.9569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5629 -1.9569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2084 -1.5768 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2084 -1.5768 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2244 -0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2244 -0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5068 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5068 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5117 -1.5683 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5117 -1.5683 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8604 -1.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8604 -1.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5003 -3.0687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5003 -3.0687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1492 -2.6926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1492 -2.6926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8008 -3.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8008 -3.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4519 -2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4519 -2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4462 -1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4462 -1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7946 -1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7946 -1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1487 -1.9471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1487 -1.9471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0214 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0214 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7880 -0.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7880 -0.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1575 -0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1575 -0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9071 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9071 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2867 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2867 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9173 0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9173 0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1682 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1682 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1117 -0.2924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1117 -0.2924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7021 -0.2628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7021 -0.2628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9500 -0.2625 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9500 -0.2625 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2000 -0.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2000 -0.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9501 0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9501 0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9478 -1.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9478 -1.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0430 -4.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0430 -4.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 6 2 0 0 0 0 | + | 1 6 2 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 7 10 2 0 0 0 0 | + | 7 10 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 15 1 0 0 0 0 | + | 9 15 1 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 11 1 0 0 0 0 | + | 15 11 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 9 16 1 0 0 0 0 | + | 9 16 1 0 0 0 0 |
| − | 17 22 2 0 0 0 0 | + | 17 22 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 24 29 2 0 0 0 0 | + | 24 29 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 12 31 1 0 0 0 0 | + | 12 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 32 35 2 0 0 0 0 | + | 32 35 2 0 0 0 0 |
| − | 2 36 1 0 0 0 0 | + | 2 36 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
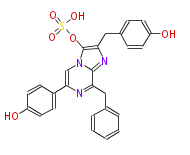
| − | ID BMCCCC--f026 | + | ID BMCCCC--f026 |
| − | NAME Luciferyl sulfate | + | NAME Luciferyl sulfate |
| − | FORMULA C26H21N3O6S | + | FORMULA C26H21N3O6S |
| − | EXACTMASS 503.1151 | + | EXACTMASS 503.1151 |
| − | AVERAGEMASS 503.5275 | + | AVERAGEMASS 503.5275 |
| − | SMILES C(=C(c(c5)ccc(O)c5)3)n(c(C(Cc(c4)cccc4)=N3)1)c(OS(O)(=O)=O)c(Cc(c2)ccc(c2)O)n1 | + | SMILES C(=C(c(c5)ccc(O)c5)3)n(c(C(Cc(c4)cccc4)=N3)1)c(OS(O)(=O)=O)c(Cc(c2)ccc(c2)O)n1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02555 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02555 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 40 0 0 0 0 0 0 0 0999 V2000
-0.3917 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3917 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2579 -4.2208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9157 -3.8375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9157 -3.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2579 -2.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5642 -2.7024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2162 -3.0755 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8502 -2.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5629 -1.9569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2084 -1.5768 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.2244 -0.8011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5068 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5117 -1.5683 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8604 -1.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5003 -3.0687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1492 -2.6926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8008 -3.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4519 -2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4462 -1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7946 -1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1487 -1.9471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0214 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7880 -0.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1575 -0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9071 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2867 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9173 0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1682 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1117 -0.2924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7021 -0.2628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9500 -0.2625 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
0.2000 -0.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9501 0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9478 -1.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0430 -4.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 6 2 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
7 10 2 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 15 1 0 0 0 0
11 10 1 0 0 0 0
5 7 1 0 0 0 0
11 12 1 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 11 1 0 0 0 0
12 13 2 0 0 0 0
9 16 1 0 0 0 0
17 22 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
16 17 1 0 0 0 0
13 23 1 0 0 0 0
24 29 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
23 24 1 0 0 0 0
27 30 1 0 0 0 0
12 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
32 35 2 0 0 0 0
2 36 1 0 0 0 0
S SKP 7
ID BMCCCC--f026
NAME Luciferyl sulfate
FORMULA C26H21N3O6S
EXACTMASS 503.1151
AVERAGEMASS 503.5275
SMILES C(=C(c(c5)ccc(O)c5)3)n(c(C(Cc(c4)cccc4)=N3)1)c(OS(O)(=O)=O)c(Cc(c2)ccc(c2)O)n1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02555
M END