Mol:FLNACAGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 35 0 0 0 0 0 0 0 0999 V2000 | + | 32 35 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6386 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6386 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0823 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0823 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4738 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4738 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4738 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4738 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0505 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0505 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6273 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6273 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6273 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6273 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0505 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0505 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6386 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6386 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0823 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0823 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0505 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0505 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4976 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4976 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4976 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4976 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0505 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0505 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6035 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6035 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6035 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6035 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2036 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2036 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0505 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0505 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0838 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0838 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0034 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.0034 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.6452 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6452 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1294 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1294 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6317 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6317 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9933 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9933 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5202 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.5202 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.0034 0.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0034 0.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2036 -1.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2036 -1.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8338 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8338 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9958 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9958 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4957 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4957 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3060 -0.0923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3060 -0.0923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9247 0.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9247 0.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 3 1 0 0 0 0 | + | 8 3 1 0 0 0 0 |
| − | 1 9 1 0 0 0 0 | + | 1 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 1 0 0 0 0 | + | 5 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 1 29 1 0 0 0 0 | + | 1 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 34 | + | M SBL 2 1 34 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 34 -1.306 -0.0923 | + | M SVB 2 34 -1.306 -0.0923 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 32 -0.9958 2.099 | + | M SVB 1 32 -0.9958 2.099 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLNACAGS0001 | + | ID FLNACAGS0001 |
| − | KNApSAcK_ID C00010237 | + | KNApSAcK_ID C00010237 |
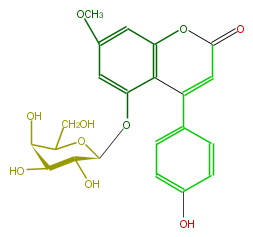
| − | NAME 5,4'-Dihydroxy-7-methoxy-4-phenylcoumarin 5-O-galactoside | + | NAME 5,4'-Dihydroxy-7-methoxy-4-phenylcoumarin 5-O-galactoside |
| − | CAS_RN 129369-35-3 | + | CAS_RN 129369-35-3 |
| − | FORMULA C22H22O10 | + | FORMULA C22H22O10 |
| − | EXACTMASS 446.121296924 | + | EXACTMASS 446.121296924 |
| − | AVERAGEMASS 446.40408 | + | AVERAGEMASS 446.40408 |
| − | SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2)O | + | SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 35 0 0 0 0 0 0 0 0999 V2000
-0.6386 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0823 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4738 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4738 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0505 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6273 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6273 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0505 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6386 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0823 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0505 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4976 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4976 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0505 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6035 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6035 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2036 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0505 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0838 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0034 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.6452 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1294 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6317 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9933 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5202 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.0034 0.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2036 -1.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8338 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9958 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4957 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3060 -0.0923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9247 0.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 3 1 0 0 0 0
1 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
19 23 1 0 0 0 0
10 19 1 0 0 0 0
1 29 1 0 0 0 0
29 30 1 0 0 0 0
25 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 34
M SMT 2 CH2OH
M SVB 2 34 -1.306 -0.0923
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 32
M SMT 1 OCH3
M SVB 1 32 -0.9958 2.099
S SKP 8
ID FLNACAGS0001
KNApSAcK_ID C00010237
NAME 5,4'-Dihydroxy-7-methoxy-4-phenylcoumarin 5-O-galactoside
CAS_RN 129369-35-3
FORMULA C22H22O10
EXACTMASS 446.121296924
AVERAGEMASS 446.40408
SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2)O
M END