Mol:FLIAA9NIN001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 35 0 0 0 0 0 0 0 0999 V2000 | + | 33 35 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0139 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0139 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0139 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0139 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5481 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5481 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0823 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0823 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0823 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0823 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5481 1.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5481 1.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6165 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6165 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1507 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1507 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1507 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1507 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6165 1.2665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6165 1.2665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3150 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3150 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7808 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7808 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3150 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3150 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7808 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7808 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2466 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2466 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2466 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2466 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7808 -1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7808 -1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6463 -0.7069 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6463 -0.7069 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6165 -0.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6165 -0.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4267 1.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4267 1.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5481 -0.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5481 -0.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7808 0.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7808 0.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2464 1.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2464 1.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2464 1.8038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2464 1.8038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7808 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7808 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7120 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7120 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1506 -0.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1506 -0.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1506 -1.2118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1506 -1.2118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6732 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6732 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1889 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1889 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5414 -2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5414 -2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7122 0.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7122 0.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4267 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4267 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 11 13 1 0 0 0 0 | + | 11 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 12 1 0 0 0 0 | + | 16 12 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 18 15 1 0 0 0 0 | + | 18 15 1 0 0 0 0 |
| − | 7 19 2 0 0 0 0 | + | 7 19 2 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 12 22 1 0 0 0 0 | + | 12 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 13 27 1 0 0 0 0 | + | 13 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 29 31 1 0 0 0 0 | + | 29 31 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 34 -5.1844 5.1026 | + | M SBV 1 34 -5.1844 5.1026 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAA9NIN001 | + | ID FLIAA9NIN001 |
| − | KNApSAcK_ID C00002561 | + | KNApSAcK_ID C00002561 |
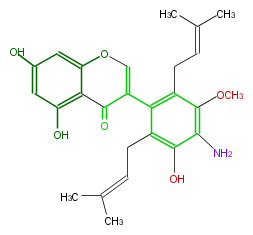
| − | NAME Piscerythramine;4'-Amino-5,7,3'-trihydroxy-5'-methoxy-2',6'-diprenylisoflavone | + | NAME Piscerythramine;4'-Amino-5,7,3'-trihydroxy-5'-methoxy-2',6'-diprenylisoflavone |
| − | CAS_RN 132923-36-5 | + | CAS_RN 132923-36-5 |
| − | FORMULA C26H29NO6 | + | FORMULA C26H29NO6 |
| − | EXACTMASS 451.199487665 | + | EXACTMASS 451.199487665 |
| − | AVERAGEMASS 451.51159999999993 | + | AVERAGEMASS 451.51159999999993 |
| − | SMILES C(=C2)(c(c(CC=C(C)C)3)c(c(O)c(c3OC)N)CC=C(C)C)C(=O)c(c(O2)1)c(O)cc(O)c1 | + | SMILES C(=C2)(c(c(CC=C(C)C)3)c(c(O)c(c3OC)N)CC=C(C)C)C(=O)c(c(O2)1)c(O)cc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 35 0 0 0 0 0 0 0 0999 V2000
-2.0139 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0139 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5481 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0823 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0823 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5481 1.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6165 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1507 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1507 0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6165 1.2665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3150 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7808 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3150 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7808 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2466 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2466 0.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7808 -1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6463 -0.7069 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-0.6165 -0.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4267 1.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5481 -0.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7808 0.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2464 1.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2464 1.8038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7808 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7120 2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1506 -0.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1506 -1.2118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6732 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1889 -1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5414 -2.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7122 0.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4267 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 2 0 0 0 0
11 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 12 1 0 0 0 0
14 17 1 0 0 0 0
18 15 1 0 0 0 0
7 19 2 0 0 0 0
20 1 1 0 0 0 0
3 21 1 0 0 0 0
12 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
24 26 1 0 0 0 0
13 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 30 1 0 0 0 0
29 31 1 0 0 0 0
16 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 34
M SMT 1 OCH3
M SBV 1 34 -5.1844 5.1026
S SKP 8
ID FLIAA9NIN001
KNApSAcK_ID C00002561
NAME Piscerythramine;4'-Amino-5,7,3'-trihydroxy-5'-methoxy-2',6'-diprenylisoflavone
CAS_RN 132923-36-5
FORMULA C26H29NO6
EXACTMASS 451.199487665
AVERAGEMASS 451.51159999999993
SMILES C(=C2)(c(c(CC=C(C)C)3)c(c(O)c(c3OC)N)CC=C(C)C)C(=O)c(c(O2)1)c(O)cc(O)c1
M END