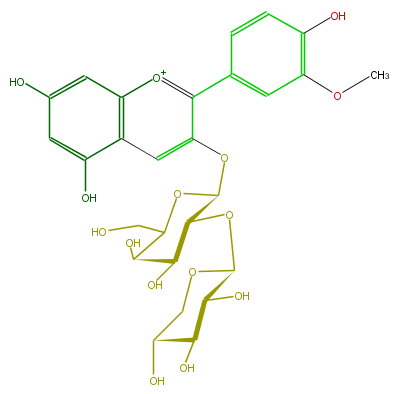
Mol:FL7AADGA0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9535 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9535 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9535 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9535 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2391 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2391 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5247 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5247 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5247 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5247 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2391 2.3981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2391 2.3981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8102 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8102 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0958 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0958 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0958 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0958 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8102 2.3981 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.8102 2.3981 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6808 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6808 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3953 2.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3953 2.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1096 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1096 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1096 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1096 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3953 3.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3953 3.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6808 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6808 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7498 3.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7498 3.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5335 2.3204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5335 2.3204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2391 -0.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2391 -0.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5544 0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5544 0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8014 2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8014 2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5335 2.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5335 2.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2769 -1.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2769 -1.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4528 -1.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4528 -1.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1951 -0.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1951 -0.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4261 0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4261 0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3982 0.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3982 0.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6559 -0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6559 -0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1910 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1910 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8877 -0.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8877 -0.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3630 -0.9099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3630 -0.9099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9081 -1.7583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9081 -1.7583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6367 -0.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6367 -0.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8847 -2.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8847 -2.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0595 -2.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0595 -2.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1468 -2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1468 -2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7316 -1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7316 -1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0936 -1.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0936 -1.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3000 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3000 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8223 -1.9761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8223 -1.9761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2820 -3.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2820 -3.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8730 -3.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8730 -3.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 37 33 1 0 0 0 0 | + | 37 33 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AADGA0003 | + | ID FL7AADGA0003 |
| − | FORMULA C27H31O15 | + | FORMULA C27H31O15 |
| − | EXACTMASS 595.166295322 | + | EXACTMASS 595.166295322 |
| − | AVERAGEMASS 595.52604 | + | AVERAGEMASS 595.52604 |
| − | SMILES c(c(c5)ccc(O)c5OC)(c2OC(C3OC(C4O)OCC(C4O)O)OC(C(C3O)O)CO)[o+1]c(c1c2)cc(O)cc1O | + | SMILES c(c(c5)ccc(O)c5OC)(c2OC(C3OC(C4O)OCC(C4O)O)OC(C(C3O)O)CO)[o+1]c(c1c2)cc(O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.9535 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9535 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2391 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5247 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5247 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2391 2.3981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8102 0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0958 1.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0958 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8102 2.3981 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.6808 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3953 2.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1096 2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1096 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3953 3.6714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6808 3.2589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7498 3.6286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5335 2.3204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2391 -0.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5544 0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8014 2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5335 2.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2769 -1.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4528 -1.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1951 -0.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4261 0.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3982 0.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6559 -0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1910 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8877 -0.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3630 -0.9099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9081 -1.7583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6367 -0.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8847 -2.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0595 -2.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1468 -2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7316 -1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0936 -1.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3000 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8223 -1.9761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2820 -3.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8730 -3.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
13 21 1 0 0 0 0
21 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 20 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
34 42 1 0 0 0 0
37 33 1 0 0 0 0
M CHG 1 10 1
S SKP 5
ID FL7AADGA0003
FORMULA C27H31O15
EXACTMASS 595.166295322
AVERAGEMASS 595.52604
SMILES c(c(c5)ccc(O)c5OC)(c2OC(C3OC(C4O)OCC(C4O)O)OC(C(C3O)O)CO)[o+1]c(c1c2)cc(O)cc1O
M END