Mol:FL7AACGL0078
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 84 91 0 0 0 0 0 0 0 0999 V2000 | + | 84 91 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2506 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2506 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2506 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2506 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6943 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6943 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1380 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1380 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1380 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1380 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6943 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6943 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5817 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5817 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0254 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0254 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0254 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0254 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5817 2.7661 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.5817 2.7661 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4693 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4693 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0977 2.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0977 2.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6647 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6647 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6647 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6647 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0977 3.7480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0977 3.7480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4693 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4693 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8067 2.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8067 2.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2315 3.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2315 3.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6943 0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6943 0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6093 1.0819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6093 1.0819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1308 -0.2322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.1308 -0.2322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.6378 0.1554 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6378 0.1554 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4282 -0.4426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4282 -0.4426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6813 -1.1406 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6813 -1.1406 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1308 -1.4585 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.1308 -1.4585 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3054 -0.8472 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.3054 -0.8472 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0440 0.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0440 0.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1960 -0.8911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1960 -0.8911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1737 -2.4110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1737 -2.4110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2059 -1.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2059 -1.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7137 0.2701 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.7137 0.2701 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.0172 -0.3218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0172 -0.3218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6259 -0.2008 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6259 -0.2008 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1766 -0.2918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1766 -0.2918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8476 0.1782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.8476 0.1782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.3029 0.0030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3029 0.0030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3952 -0.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3952 -0.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8557 -0.8063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8557 -0.8063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1298 -0.5941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1298 -0.5941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0977 4.4025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0977 4.4025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8436 -2.6260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8436 -2.6260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8530 0.5651 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.8530 0.5651 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.4819 0.0751 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.4819 0.0751 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.9474 0.2830 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9474 0.2830 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.4316 0.2886 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.4316 0.2886 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.8064 0.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8064 0.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2740 0.4166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.2740 0.4166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.3668 0.8618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3668 0.8618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8323 -0.3861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8323 -0.3861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6411 -0.2311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6411 -0.2311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6446 0.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6446 0.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3058 1.3955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3058 1.3955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1815 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1815 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8537 -3.7789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8537 -3.7789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7483 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7483 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0723 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0723 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6992 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6992 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0117 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0117 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6367 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6367 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9492 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9492 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6367 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6367 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0117 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0117 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5734 -3.7726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5734 -3.7726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8324 0.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8324 0.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7634 0.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7634 0.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3389 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3389 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8701 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8701 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4500 1.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4500 1.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9187 1.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9187 1.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0367 2.3183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0367 2.3183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6454 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6454 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7579 3.1778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7579 3.1778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2617 3.5942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2617 3.5942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6530 3.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6530 3.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5405 2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5405 2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3741 4.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3741 4.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3994 -2.8430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3994 -2.8430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0752 -2.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0752 -2.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4085 4.0440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4085 4.0440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0664 4.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0664 4.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2922 -4.5339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2922 -4.5339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5777 -4.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5777 -4.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3665 3.3993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3665 3.3993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9985 2.8690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9985 2.8690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 15 40 1 0 0 0 0 | + | 15 40 1 0 0 0 0 |
| − | 30 41 1 0 0 0 0 | + | 30 41 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 19 1 0 0 0 0 | + | 45 19 1 0 0 0 0 |
| − | 41 53 1 0 0 0 0 | + | 41 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 53 55 1 0 0 0 0 | + | 53 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 62 2 0 0 0 0 | + | 61 62 2 0 0 0 0 |
| − | 62 57 1 0 0 0 0 | + | 62 57 1 0 0 0 0 |
| − | 60 63 1 0 0 0 0 | + | 60 63 1 0 0 0 0 |
| − | 35 64 1 0 0 0 0 | + | 35 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 66 68 1 0 0 0 0 | + | 66 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 66 65 1 0 0 0 0 | + | 66 65 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 72 73 2 0 0 0 0 | + | 72 73 2 0 0 0 0 |
| − | 73 74 1 0 0 0 0 | + | 73 74 1 0 0 0 0 |
| − | 74 75 2 0 0 0 0 | + | 74 75 2 0 0 0 0 |
| − | 75 70 1 0 0 0 0 | + | 75 70 1 0 0 0 0 |
| − | 73 76 1 0 0 0 0 | + | 73 76 1 0 0 0 0 |
| − | 59 77 1 0 0 0 0 | + | 59 77 1 0 0 0 0 |
| − | 77 78 1 0 0 0 0 | + | 77 78 1 0 0 0 0 |
| − | 74 79 1 0 0 0 0 | + | 74 79 1 0 0 0 0 |
| − | 79 80 1 0 0 0 0 | + | 79 80 1 0 0 0 0 |
| − | 61 81 1 0 0 0 0 | + | 61 81 1 0 0 0 0 |
| − | 81 82 1 0 0 0 0 | + | 81 82 1 0 0 0 0 |
| − | 72 83 1 0 0 0 0 | + | 72 83 1 0 0 0 0 |
| − | 83 84 1 0 0 0 0 | + | 83 84 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 51 52 | + | M SAL 5 2 51 52 |
| − | M SBL 5 1 55 | + | M SBL 5 1 55 |
| − | M SMT 5 CH2OH | + | M SMT 5 CH2OH |
| − | M SVB 5 55 -4.7389 0.9187 | + | M SVB 5 55 -4.7389 0.9187 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 83 84 | + | M SAL 4 2 83 84 |
| − | M SBL 4 1 90 | + | M SBL 4 1 90 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 90 4.6524 4.2987 | + | M SVB 4 90 4.6524 4.2987 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 81 82 | + | M SAL 3 2 81 82 |
| − | M SBL 3 1 88 | + | M SBL 3 1 88 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 88 -4.2922 -4.5339 | + | M SVB 3 88 -4.2922 -4.5339 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 79 80 | + | M SAL 2 2 79 80 |
| − | M SBL 2 1 86 | + | M SBL 2 1 86 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 86 2.6122 4.5936 | + | M SVB 2 86 2.6122 4.5936 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 77 78 | + | M SAL 1 2 77 78 |
| − | M SBL 1 1 84 | + | M SBL 1 1 84 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 84 -3.8295 -2.3975 | + | M SVB 1 84 -3.8295 -2.3975 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0078 | + | ID FL7AACGL0078 |
| − | KNApSAcK_ID C00006854 | + | KNApSAcK_ID C00006854 |
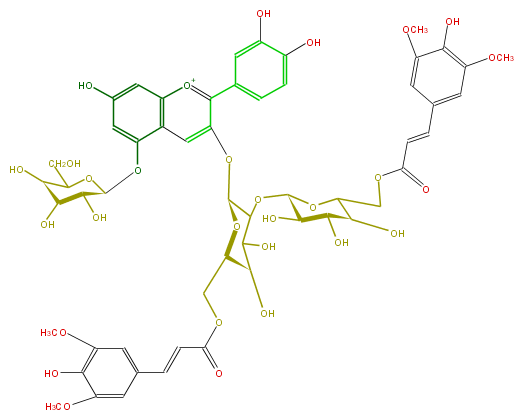
| − | NAME Cyanidin 3-(6'',6'''-disinapylsophoroside)-5-glucoside | + | NAME Cyanidin 3-(6'',6'''-disinapylsophoroside)-5-glucoside |
| − | CAS_RN 110202-93-2 | + | CAS_RN 110202-93-2 |
| − | FORMULA C55H61O29 | + | FORMULA C55H61O29 |
| − | EXACTMASS 1185.32985099 | + | EXACTMASS 1185.32985099 |
| − | AVERAGEMASS 1186.0554399999999 | + | AVERAGEMASS 1186.0554399999999 |
| − | SMILES Oc(c(OC)1)c(OC)cc(C=CC(OC[C@@H](O4)[C@@H](C(C([C@H](Oc(c7)c(c(c8)ccc(O)c(O)8)[o+1]c(c57)cc(cc5O[C@H](O6)[C@@H](O)[C@@H](O)[C@H](C(CO)6)O)O)4)O[C@H]([C@H](O)2)OC(COC(=O)C=Cc(c3)cc(c(O)c3OC)OC)[C@H]([C@H]2O)O)O)O)=O)c1 | + | SMILES Oc(c(OC)1)c(OC)cc(C=CC(OC[C@@H](O4)[C@@H](C(C([C@H](Oc(c7)c(c(c8)ccc(O)c(O)8)[o+1]c(c57)cc(cc5O[C@H](O6)[C@@H](O)[C@@H](O)[C@H](C(CO)6)O)O)4)O[C@H]([C@H](O)2)OC(COC(=O)C=Cc(c3)cc(c(O)c3OC)OC)[C@H]([C@H]2O)O)O)O)=O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
84 91 0 0 0 0 0 0 0 0999 V2000
-3.2506 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2506 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6943 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1380 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1380 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6943 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5817 1.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0254 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0254 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5817 2.7661 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.4693 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0977 2.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6647 2.7660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6647 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0977 3.7480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4693 3.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8067 2.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2315 3.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6943 0.8393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6093 1.0819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1308 -0.2322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.6378 0.1554 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4282 -0.4426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6813 -1.1406 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1308 -1.4585 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3054 -0.8472 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0440 0.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1960 -0.8911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1737 -2.4110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2059 -1.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7137 0.2701 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.0172 -0.3218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6259 -0.2008 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1766 -0.2918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8476 0.1782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.3029 0.0030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3952 -0.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8557 -0.8063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1298 -0.5941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0977 4.4025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8436 -2.6260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8530 0.5651 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.4819 0.0751 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.9474 0.2830 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.4316 0.2886 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.8064 0.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2740 0.4166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.3668 0.8618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8323 -0.3861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6411 -0.2311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6446 0.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3058 1.3955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1815 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8537 -3.7789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7483 -3.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0723 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6992 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0117 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6367 -3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9492 -3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6367 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0117 -4.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5734 -3.7726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8324 0.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7634 0.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3389 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8701 0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4500 1.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9187 1.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0367 2.3183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6454 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7579 3.1778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2617 3.5942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6530 3.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5405 2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3741 4.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3994 -2.8430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0752 -2.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4085 4.0440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0664 4.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2922 -4.5339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5777 -4.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3665 3.3993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9985 2.8690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
27 31 1 0 0 0 0
22 20 1 0 0 0 0
15 40 1 0 0 0 0
30 41 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
47 51 1 0 0 0 0
51 52 1 0 0 0 0
45 19 1 0 0 0 0
41 53 1 0 0 0 0
53 54 2 0 0 0 0
53 55 1 0 0 0 0
55 56 2 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
60 61 1 0 0 0 0
61 62 2 0 0 0 0
62 57 1 0 0 0 0
60 63 1 0 0 0 0
35 64 1 0 0 0 0
64 65 1 0 0 0 0
66 67 2 0 0 0 0
66 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
66 65 1 0 0 0 0
70 71 2 0 0 0 0
71 72 1 0 0 0 0
72 73 2 0 0 0 0
73 74 1 0 0 0 0
74 75 2 0 0 0 0
75 70 1 0 0 0 0
73 76 1 0 0 0 0
59 77 1 0 0 0 0
77 78 1 0 0 0 0
74 79 1 0 0 0 0
79 80 1 0 0 0 0
61 81 1 0 0 0 0
81 82 1 0 0 0 0
72 83 1 0 0 0 0
83 84 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 51 52
M SBL 5 1 55
M SMT 5 CH2OH
M SVB 5 55 -4.7389 0.9187
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 83 84
M SBL 4 1 90
M SMT 4 OCH3
M SVB 4 90 4.6524 4.2987
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 81 82
M SBL 3 1 88
M SMT 3 OCH3
M SVB 3 88 -4.2922 -4.5339
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 79 80
M SBL 2 1 86
M SMT 2 OCH3
M SVB 2 86 2.6122 4.5936
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 77 78
M SBL 1 1 84
M SMT 1 OCH3
M SVB 1 84 -3.8295 -2.3975
S SKP 8
ID FL7AACGL0078
KNApSAcK_ID C00006854
NAME Cyanidin 3-(6'',6'''-disinapylsophoroside)-5-glucoside
CAS_RN 110202-93-2
FORMULA C55H61O29
EXACTMASS 1185.32985099
AVERAGEMASS 1186.0554399999999
SMILES Oc(c(OC)1)c(OC)cc(C=CC(OC[C@@H](O4)[C@@H](C(C([C@H](Oc(c7)c(c(c8)ccc(O)c(O)8)[o+1]c(c57)cc(cc5O[C@H](O6)[C@@H](O)[C@@H](O)[C@H](C(CO)6)O)O)4)O[C@H]([C@H](O)2)OC(COC(=O)C=Cc(c3)cc(c(O)c3OC)OC)[C@H]([C@H]2O)O)O)O)=O)c1
M END