Mol:FL7AACGL0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6687 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6687 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6687 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6687 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1682 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1682 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6678 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6678 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6678 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6678 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1682 0.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1682 0.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1673 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1673 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3331 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3331 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3331 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3331 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1673 0.8790 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.1673 0.8790 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8332 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8332 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3403 0.5860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3403 0.5860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8473 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8473 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8473 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8473 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3403 1.7570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3403 1.7570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8332 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8332 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7444 -0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7444 -0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8994 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8994 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5074 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5074 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2613 -0.6573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2613 -0.6573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0197 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0197 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4118 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4118 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6579 -0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6579 -0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4259 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4259 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9265 0.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9265 0.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9857 -0.3475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9857 -0.3475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1688 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1688 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1682 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1682 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3534 1.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3534 1.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6899 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6899 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2257 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2257 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6554 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6554 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9990 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9990 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4632 -0.8380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4632 -0.8380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0336 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0336 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2508 -1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2508 -1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9411 -1.6273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9411 -1.6273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2006 -1.7570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2006 -1.7570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3534 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3534 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4539 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4539 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2508 -0.9708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2508 -0.9708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1302 -0.7647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1302 -0.7647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9271 -0.5511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9271 -0.5511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 8 17 1 0 0 0 0 | + | 8 17 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 19 17 1 0 0 0 0 | + | 19 17 1 0 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 14 29 1 0 0 0 0 | + | 14 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 13 39 1 0 0 0 0 | + | 13 39 1 0 0 0 0 |
| − | 21 40 1 0 0 0 0 | + | 21 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -5.4482 4.4385 | + | M SBV 1 44 -5.4482 4.4385 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 46 -5.9790 4.7216 | + | M SBV 2 46 -5.9790 4.7216 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0004 | + | ID FL7AACGL0004 |
| − | KNApSAcK_ID C00002378 | + | KNApSAcK_ID C00002378 |
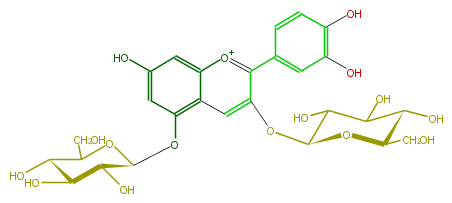
| − | NAME Cyanin;Cyanidin 3,5-diglucoside | + | NAME Cyanin;Cyanidin 3,5-diglucoside |
| − | CAS_RN 2611-67-8 | + | CAS_RN 2611-67-8 |
| − | FORMULA C27H31O16 | + | FORMULA C27H31O16 |
| − | EXACTMASS 611.161209944 | + | EXACTMASS 611.161209944 |
| − | AVERAGEMASS 611.52544 | + | AVERAGEMASS 611.52544 |
| − | SMILES Oc(c(O)1)ccc(c(c4OC(O5)C(C(O)C(C5CO)O)O)[o+1]c(c3)c(c4)c(cc3O)OC(O2)C(C(C(O)C2CO)O)O)c1 | + | SMILES Oc(c(O)1)ccc(c(c4OC(O5)C(C(O)C(C5CO)O)O)[o+1]c(c3)c(c4)c(cc3O)OC(O2)C(C(C(O)C2CO)O)O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.6687 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6687 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1682 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6678 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6678 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1682 0.8790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1673 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3331 0.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3331 0.5900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1673 0.8790 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.8332 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3403 0.5860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8473 0.8788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8473 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3403 1.7570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8332 1.4643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7444 -0.5856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8994 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5074 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2613 -0.6573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0197 -0.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4118 -0.1937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6579 -0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4259 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9265 0.0562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9857 -0.3475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1688 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1682 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3534 1.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6899 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2257 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6554 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9990 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4632 -0.8380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0336 -1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2508 -1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9411 -1.6273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2006 -1.7570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3534 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4539 -0.7573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2508 -0.9708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1302 -0.7647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9271 -0.5511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
8 17 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
19 17 1 0 0 0 0
18 24 1 0 0 0 0
23 25 1 0 0 0 0
22 26 1 0 0 0 0
1 27 1 0 0 0 0
3 28 1 0 0 0 0
14 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
33 28 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
13 39 1 0 0 0 0
21 40 1 0 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -5.4482 4.4385
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 ^CH2OH
M SBV 2 46 -5.9790 4.7216
S SKP 8
ID FL7AACGL0004
KNApSAcK_ID C00002378
NAME Cyanin;Cyanidin 3,5-diglucoside
CAS_RN 2611-67-8
FORMULA C27H31O16
EXACTMASS 611.161209944
AVERAGEMASS 611.52544
SMILES Oc(c(O)1)ccc(c(c4OC(O5)C(C(O)C(C5CO)O)O)[o+1]c(c3)c(c4)c(cc3O)OC(O2)C(C(C(O)C2CO)O)O)c1
M END