Mol:FL63BINS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2456 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2456 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2456 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2456 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7070 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7070 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1685 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1685 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1685 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1685 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7070 1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7070 1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6300 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6300 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 0.4739 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.0915 0.4739 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 1.0957 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.0915 1.0957 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.6300 1.4066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6300 1.4066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7838 1.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7838 1.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3881 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3881 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9328 1.0581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9328 1.0581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4775 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4775 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4775 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4775 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9328 2.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9328 2.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3881 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3881 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3304 -0.0760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3304 -0.0760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4775 2.0016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4775 2.0016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3304 -0.7814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3304 -0.7814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 -1.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 -1.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9966 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9966 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9966 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9966 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5925 -2.1981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5925 -2.1981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1884 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1884 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1884 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1884 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5925 -0.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5925 -0.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5925 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5925 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7838 -2.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7838 -2.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7838 -0.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7838 -0.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2049 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2049 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6359 3.7879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6359 3.7879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3435 0.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3435 0.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3435 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3435 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1524 0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1524 0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4379 -0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4379 -0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 6 0 0 0 | + | 8 18 1 6 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 18 20 1 0 0 0 0 | + | 18 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 14 33 1 0 0 0 0 | + | 14 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 3 35 1 0 0 0 0 | + | 3 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 35 36 | + | M SAL 3 2 35 36 |
| − | M SBL 3 1 38 | + | M SBL 3 1 38 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 38 -2.1524 0.0076 | + | M SVB 3 38 -2.1524 0.0076 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 33 34 | + | M SAL 2 2 33 34 |
| − | M SBL 2 1 36 | + | M SBL 2 1 36 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 36 1.6645 1.1663 | + | M SVB 2 36 1.6645 1.1663 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 34 1.2049 2.8856 | + | M SVB 1 34 1.2049 2.8856 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63BINS0001 | + | ID FL63BINS0001 |
| − | KNApSAcK_ID C00008893 | + | KNApSAcK_ID C00008893 |
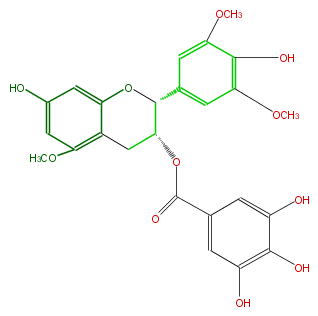
| − | NAME Epigallocatechin 5,3',5'-trimethyl ether 3-O-gallate | + | NAME Epigallocatechin 5,3',5'-trimethyl ether 3-O-gallate |
| − | CAS_RN 148707-36-2 | + | CAS_RN 148707-36-2 |
| − | FORMULA C25H24O11 | + | FORMULA C25H24O11 |
| − | EXACTMASS 500.13186161 | + | EXACTMASS 500.13186161 |
| − | AVERAGEMASS 500.45146 | + | AVERAGEMASS 500.45146 |
| − | SMILES O([C@H](C4)[C@H](Oc(c34)cc(cc(OC)3)O)c(c2)cc(c(O)c(OC)2)OC)C(=O)c(c1)cc(c(c1O)O)O | + | SMILES O([C@H](C4)[C@H](Oc(c34)cc(cc(OC)3)O)c(c2)cc(c(O)c(OC)2)OC)C(=O)c(c1)cc(c(c1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-2.2456 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2456 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7070 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1685 0.4739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1685 1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7070 1.4066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6300 0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0915 0.4739 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.0915 1.0957 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.6300 1.4066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7838 1.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3881 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9328 1.0581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4775 1.3726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4775 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9328 2.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3881 2.0016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3304 -0.0760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4775 2.0016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3304 -0.7814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 -1.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9966 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9966 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5925 -2.1981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1884 -1.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1884 -1.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5925 -0.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5925 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7838 -2.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7838 -0.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2049 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6359 3.7879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3435 0.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3435 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1524 0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4379 -0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 6 0 0 0
15 19 1 0 0 0 0
18 20 1 0 0 0 0
20 21 2 0 0 0 0
20 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
24 28 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
16 31 1 0 0 0 0
31 32 1 0 0 0 0
14 33 1 0 0 0 0
33 34 1 0 0 0 0
3 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 35 36
M SBL 3 1 38
M SMT 3 OCH3
M SVB 3 38 -2.1524 0.0076
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 33 34
M SBL 2 1 36
M SMT 2 OCH3
M SVB 2 36 1.6645 1.1663
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 34
M SMT 1 OCH3
M SVB 1 34 1.2049 2.8856
S SKP 8
ID FL63BINS0001
KNApSAcK_ID C00008893
NAME Epigallocatechin 5,3',5'-trimethyl ether 3-O-gallate
CAS_RN 148707-36-2
FORMULA C25H24O11
EXACTMASS 500.13186161
AVERAGEMASS 500.45146
SMILES O([C@H](C4)[C@H](Oc(c34)cc(cc(OC)3)O)c(c2)cc(c(O)c(OC)2)OC)C(=O)c(c1)cc(c(c1O)O)O
M END