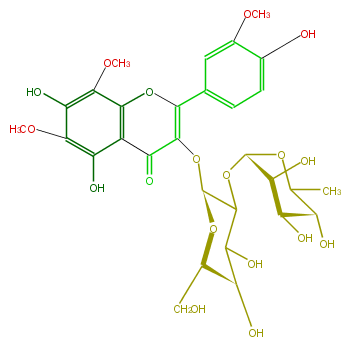
Mol:FL5FGCGL0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2454 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2454 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2454 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2454 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6891 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6891 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1328 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1328 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1328 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1328 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6891 1.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6891 1.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5765 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5765 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0202 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0202 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0202 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0202 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5765 1.6006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5765 1.6006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5765 -0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5765 -0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1028 1.2731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1028 1.2731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6698 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6698 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6698 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6698 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1028 2.5825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1028 2.5825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8015 1.6005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8015 1.6005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5358 2.7552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5358 2.7552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3739 0.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3739 0.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1625 -0.7790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.1625 -0.7790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.4911 -0.3914 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4911 -0.3914 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7042 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7042 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4911 -1.8868 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4911 -1.8868 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.1625 -2.2745 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1625 -2.2745 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.9495 -1.5290 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9495 -1.5290 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9696 -0.0590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9696 -0.0590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4812 -1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4812 -1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4983 -3.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4983 -3.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0397 -0.8809 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.0397 -0.8809 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.7651 -0.8809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7651 -0.8809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2437 -0.3765 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2437 -0.3765 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.0655 0.3309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0655 0.3309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3400 0.3309 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3400 0.3309 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8614 -0.1735 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8614 -0.1735 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5285 0.2116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5285 0.2116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4805 -1.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4805 -1.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9167 -1.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9167 -1.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9599 -0.3765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9599 -0.3765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6891 -0.3262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6891 -0.3262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4111 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4111 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9746 3.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9746 3.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3862 3.1586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3862 3.1586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8276 4.0559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8276 4.0559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0272 -2.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0272 -2.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0270 -2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0270 -2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9332 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9332 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6476 0.4385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6476 0.4385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 34 27 1 0 0 0 0 | + | 34 27 1 0 0 0 0 |
| − | 3 40 1 0 0 0 0 | + | 3 40 1 0 0 0 0 |
| − | 6 41 1 0 0 0 0 | + | 6 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 24 45 1 0 0 0 0 | + | 24 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 2 47 1 0 0 0 0 | + | 2 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 45 46 | + | M SAL 4 2 45 46 |
| − | M SBL 4 1 49 | + | M SBL 4 1 49 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 49 0.9652 -2.5753 | + | M SVB 4 49 0.9652 -2.5753 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 47 48 | + | M SAL 3 2 47 48 |
| − | M SBL 3 1 51 | + | M SBL 3 1 51 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 51 -2.9599 0.7285 | + | M SVB 3 51 -2.9599 0.7285 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 47 1.3862 3.1586 | + | M SVB 2 47 1.3862 3.1586 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 45 -1.4111 2.1736 | + | M SVB 1 45 -1.4111 2.1736 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FGCGL0002 | + | ID FL5FGCGL0002 |
| − | KNApSAcK_ID C00005785 | + | KNApSAcK_ID C00005785 |
| − | NAME Limocitrol 3-neohesperidoside | + | NAME Limocitrol 3-neohesperidoside |
| − | CAS_RN 122327-80-4 | + | CAS_RN 122327-80-4 |
| − | FORMULA C30H36O18 | + | FORMULA C30H36O18 |
| − | EXACTMASS 684.190164348 | + | EXACTMASS 684.190164348 |
| − | AVERAGEMASS 684.59604 | + | AVERAGEMASS 684.59604 |
| − | SMILES CC([C@@H](O)5)O[C@@H]([C@@H](O)[C@H](O)5)OC(C(O)1)[C@H](OC(=C2c(c4)cc(c(O)c4)OC)C(=O)c(c3O)c(c(c(c3OC)O)OC)O2)O[C@H](CO)[C@H](O)1 | + | SMILES CC([C@@H](O)5)O[C@@H]([C@@H](O)[C@H](O)5)OC(C(O)1)[C@H](OC(=C2c(c4)cc(c(O)c4)OC)C(=O)c(c3O)c(c(c(c3OC)O)OC)O2)O[C@H](CO)[C@H](O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
-2.2454 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2454 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6891 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1328 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1328 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6891 1.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5765 0.3159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0202 0.6371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0202 1.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5765 1.6006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5765 -0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1028 1.2731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6698 1.6005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6698 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1028 2.5825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 2.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8015 1.6005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5358 2.7552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3739 0.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1625 -0.7790 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.4911 -0.3914 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7042 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4911 -1.8868 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.1625 -2.2745 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.9495 -1.5290 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9696 -0.0590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4812 -1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4983 -3.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0397 -0.8809 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.7651 -0.8809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2437 -0.3765 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.0655 0.3309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3400 0.3309 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8614 -0.1735 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5285 0.2116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4805 -1.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9167 -1.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9599 -0.3765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6891 -0.3262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4111 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9746 3.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3862 3.1586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8276 4.0559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0272 -2.7420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0270 -2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9332 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6476 0.4385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
35 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
34 27 1 0 0 0 0
3 40 1 0 0 0 0
6 41 1 0 0 0 0
41 42 1 0 0 0 0
16 43 1 0 0 0 0
43 44 1 0 0 0 0
24 45 1 0 0 0 0
45 46 1 0 0 0 0
2 47 1 0 0 0 0
47 48 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 45 46
M SBL 4 1 49
M SMT 4 CH2OH
M SVB 4 49 0.9652 -2.5753
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 47 48
M SBL 3 1 51
M SMT 3 OCH3
M SVB 3 51 -2.9599 0.7285
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 OCH3
M SVB 2 47 1.3862 3.1586
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 OCH3
M SVB 1 45 -1.4111 2.1736
S SKP 8
ID FL5FGCGL0002
KNApSAcK_ID C00005785
NAME Limocitrol 3-neohesperidoside
CAS_RN 122327-80-4
FORMULA C30H36O18
EXACTMASS 684.190164348
AVERAGEMASS 684.59604
SMILES CC([C@@H](O)5)O[C@@H]([C@@H](O)[C@H](O)5)OC(C(O)1)[C@H](OC(=C2c(c4)cc(c(O)c4)OC)C(=O)c(c3O)c(c(c(c3OC)O)OC)O2)O[C@H](CO)[C@H](O)1
M END