Mol:FL5FFCNS0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 27 29 0 0 0 0 0 0 0 0999 V2000 | + | 27 29 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0624 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0624 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0624 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0624 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5061 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5061 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9498 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9498 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9498 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9498 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5061 0.1844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5061 0.1844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3935 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3935 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1628 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1628 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1628 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1628 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3935 0.1844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3935 0.1844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3935 -1.6012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3935 -1.6012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7189 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7189 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2859 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2859 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8528 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8528 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8528 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8528 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2859 1.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2859 1.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7189 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7189 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5061 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5061 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8528 0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8528 0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2281 0.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2281 0.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7916 1.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7916 1.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0288 -1.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0288 -1.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8949 -1.7790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8949 -1.7790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4197 0.4820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4197 0.4820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9197 1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9197 1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5693 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5693 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0107 2.6397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0107 2.6397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 8 22 1 0 0 0 0 | + | 8 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 1 24 1 0 0 0 0 | + | 1 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 16 26 1 0 0 0 0 | + | 16 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 26 27 | + | M SAL 4 2 26 27 |
| − | M SBL 4 1 28 | + | M SBL 4 1 28 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 28 1.5693 1.7424 | + | M SVB 4 28 1.5693 1.7424 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 24 25 | + | M SAL 3 2 24 25 |
| − | M SBL 3 1 26 | + | M SBL 3 1 26 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 26 -2.4197 0.482 | + | M SVB 3 26 -2.4197 0.482 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 22 23 | + | M SAL 2 2 22 23 |
| − | M SBL 2 1 24 | + | M SBL 2 1 24 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 24 0.3617 -0.9854 | + | M SVB 2 24 0.3617 -0.9854 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 20 21 | + | M SAL 1 2 20 21 |
| − | M SBL 1 1 22 | + | M SBL 1 1 22 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 22 -1.2281 0.7574 | + | M SVB 1 22 -1.2281 0.7574 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FFCNS0017 | + | ID FL5FFCNS0017 |
| − | KNApSAcK_ID C00004737 | + | KNApSAcK_ID C00004737 |
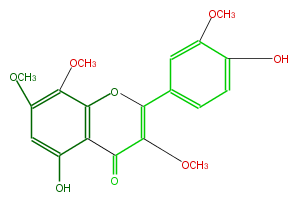
| − | NAME Ternatin;5,4'-Dihydroxy-3,7,8,3'-tetramethoxyflavone;Gossypetin 3,7,8,3'-tetramethyl ether;5-Hydroxy-2-(4-hydroxy-3-methoxyphenyl)-3,7,8-trimethoxy-4H-1-benzopyran-4-one | + | NAME Ternatin;5,4'-Dihydroxy-3,7,8,3'-tetramethoxyflavone;Gossypetin 3,7,8,3'-tetramethyl ether;5-Hydroxy-2-(4-hydroxy-3-methoxyphenyl)-3,7,8-trimethoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 571-71-1 | + | CAS_RN 571-71-1 |
| − | FORMULA C19H18O8 | + | FORMULA C19H18O8 |
| − | EXACTMASS 374.100167552 | + | EXACTMASS 374.100167552 |
| − | AVERAGEMASS 374.34142 | + | AVERAGEMASS 374.34142 |
| − | SMILES c(c1OC)(OC)cc(c(C2=O)c(OC(c(c3)cc(OC)c(O)c3)=C2OC)1)O | + | SMILES c(c1OC)(OC)cc(c(C2=O)c(OC(c(c3)cc(OC)c(O)c3)=C2OC)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
27 29 0 0 0 0 0 0 0 0999 V2000
-2.0624 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0624 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5061 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9498 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9498 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5061 0.1844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3935 -1.1003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1628 -0.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1628 -0.1368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3935 0.1844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3935 -1.6012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7189 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2859 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8528 0.1843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8528 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2859 1.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7189 0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5061 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8528 0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2281 0.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7916 1.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0288 -1.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8949 -1.7790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4197 0.4820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9197 1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5693 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0107 2.6397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
6 20 1 0 0 0 0
20 21 1 0 0 0 0
8 22 1 0 0 0 0
22 23 1 0 0 0 0
1 24 1 0 0 0 0
24 25 1 0 0 0 0
16 26 1 0 0 0 0
26 27 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 26 27
M SBL 4 1 28
M SMT 4 OCH3
M SVB 4 28 1.5693 1.7424
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 24 25
M SBL 3 1 26
M SMT 3 OCH3
M SVB 3 26 -2.4197 0.482
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 22 23
M SBL 2 1 24
M SMT 2 OCH3
M SVB 2 24 0.3617 -0.9854
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 20 21
M SBL 1 1 22
M SMT 1 OCH3
M SVB 1 22 -1.2281 0.7574
S SKP 8
ID FL5FFCNS0017
KNApSAcK_ID C00004737
NAME Ternatin;5,4'-Dihydroxy-3,7,8,3'-tetramethoxyflavone;Gossypetin 3,7,8,3'-tetramethyl ether;5-Hydroxy-2-(4-hydroxy-3-methoxyphenyl)-3,7,8-trimethoxy-4H-1-benzopyran-4-one
CAS_RN 571-71-1
FORMULA C19H18O8
EXACTMASS 374.100167552
AVERAGEMASS 374.34142
SMILES c(c1OC)(OC)cc(c(C2=O)c(OC(c(c3)cc(OC)c(O)c3)=C2OC)1)O
M END