Mol:FL5FFCGS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.6028 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6028 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8884 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8884 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8883 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8883 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6028 1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6028 1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3173 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3173 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3173 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3173 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1739 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1739 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4594 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4594 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2551 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2551 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2551 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2551 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4594 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4594 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1739 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1739 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9695 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9695 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6840 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6840 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6840 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6840 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9695 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9695 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4594 -1.6426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4594 -1.6426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3985 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3985 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8883 -0.9356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8883 -0.9356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9695 -1.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9695 -1.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0318 1.9519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0318 1.9519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6028 2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6028 2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9695 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9695 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6601 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6601 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5329 -0.5634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5329 -0.5634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5486 -1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5486 -1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1359 -2.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1359 -2.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3376 -2.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3376 -2.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5441 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5441 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9567 -1.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9567 -1.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7551 -1.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7551 -1.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9120 -1.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9120 -1.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3919 -0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3919 -0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0318 -2.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0318 -2.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5846 -2.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5846 -2.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5091 -2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5091 -2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 28 36 1 0 0 0 0 | + | 28 36 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FFCGS0025 | + | ID FL5FFCGS0025 |
| − | KNApSAcK_ID C00013960 | + | KNApSAcK_ID C00013960 |
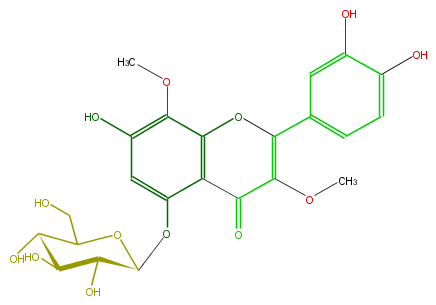
| − | NAME Gossypetin 3,8-dimethyl ether 5-glucoside;2-(3,4-Dihydroxyphenyl)-5-(beta-D-glucopyranosyloxy)-7-hydroxy-3,8-dimethoxy-4H-1-benzopyran-4-one | + | NAME Gossypetin 3,8-dimethyl ether 5-glucoside;2-(3,4-Dihydroxyphenyl)-5-(beta-D-glucopyranosyloxy)-7-hydroxy-3,8-dimethoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 637774-69-7 | + | CAS_RN 637774-69-7 |
| − | FORMULA C23H24O13 | + | FORMULA C23H24O13 |
| − | EXACTMASS 508.121690854 | + | EXACTMASS 508.121690854 |
| − | AVERAGEMASS 508.42886 | + | AVERAGEMASS 508.42886 |
| − | SMILES c(c1O)(c(O3)c(C(=O)C(OC)=C3c(c4)ccc(c4O)O)c(OC(C(O)2)OC(CO)C(C2O)O)c1)OC | + | SMILES c(c1O)(c(O3)c(C(=O)C(OC)=C3c(c4)ccc(c4O)O)c(OC(C(O)2)OC(CO)C(C2O)O)c1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
2.6028 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8884 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8883 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6028 1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3173 1.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3173 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1739 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4594 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2551 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2551 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4594 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1739 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9695 0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6840 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6840 -0.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9695 -0.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4594 -1.6426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3985 0.7144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8883 -0.9356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9695 -1.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0318 1.9519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6028 2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9695 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6601 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5329 -0.5634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5486 -1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1359 -2.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3376 -2.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5441 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9567 -1.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7551 -1.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9120 -1.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3919 -0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0318 -2.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5846 -2.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5091 -2.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
19 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
26 34 1 0 0 0 0
27 35 1 0 0 0 0
28 36 1 0 0 0 0
29 20 1 0 0 0 0
S SKP 8
ID FL5FFCGS0025
KNApSAcK_ID C00013960
NAME Gossypetin 3,8-dimethyl ether 5-glucoside;2-(3,4-Dihydroxyphenyl)-5-(beta-D-glucopyranosyloxy)-7-hydroxy-3,8-dimethoxy-4H-1-benzopyran-4-one
CAS_RN 637774-69-7
FORMULA C23H24O13
EXACTMASS 508.121690854
AVERAGEMASS 508.42886
SMILES c(c1O)(c(O3)c(C(=O)C(OC)=C3c(c4)ccc(c4O)O)c(OC(C(O)2)OC(CO)C(C2O)O)c1)OC
M END