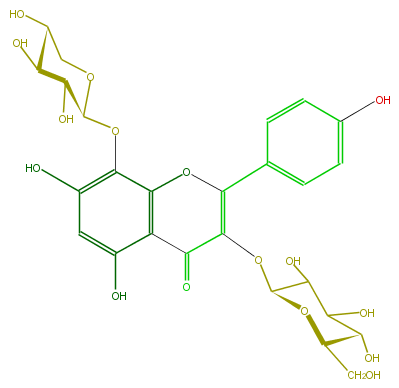
Mol:FL5FFAGL0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3689 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3689 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3688 -0.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3688 -0.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6542 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6542 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9396 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9396 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9397 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9397 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6542 0.8914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6542 0.8914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2250 -0.7588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2250 -0.7588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4895 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4895 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4895 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4895 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2250 0.8914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2250 0.8914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2248 -1.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2248 -1.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3895 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3895 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1176 0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1176 0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8460 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8460 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8460 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8460 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1176 2.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1176 2.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3894 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3894 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6541 -1.5837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6541 -1.5837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6255 2.3411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6255 2.3411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2132 -0.8725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2132 -0.8725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2223 0.9716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2223 0.9716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6542 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6542 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0073 3.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0073 3.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1928 2.9103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1928 2.9103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6629 2.6984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6629 2.6984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2919 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2919 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1435 2.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1435 2.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7292 3.1178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7292 3.1178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6255 3.4837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6255 3.4837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7068 1.9544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7068 1.9544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2081 -1.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2081 -1.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4616 -1.5154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4616 -1.5154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1372 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1372 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5157 -2.5695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5157 -2.5695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2622 -2.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2622 -2.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5867 -1.9940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5867 -1.9940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8427 -0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8427 -0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3251 -1.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3251 -1.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4174 -2.8280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4174 -2.8280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5465 4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5465 4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0687 -3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0687 -3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1412 -4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1412 -4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 20 32 1 0 0 0 0 | + | 20 32 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.5530 0.5980 | + | M SBV 1 46 -0.5530 0.5980 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FFAGL0002 | + | ID FL5FFAGL0002 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES C(C(C1O)OC(OC(C2=O)=C(c(c5)ccc(O)c5)Oc(c(OC(C4O)OCC(C4O)O)3)c2c(O)cc3O)C(O)C(O)1)O | + | SMILES C(C(C1O)OC(OC(C2=O)=C(c(c5)ccc(O)c5)Oc(c(OC(C4O)OCC(C4O)O)3)c2c(O)cc3O)C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.3689 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3688 -0.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6542 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9396 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9397 0.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6542 0.8914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2250 -0.7588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4895 -0.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4895 0.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2250 0.8914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2248 -1.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3895 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1176 0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8460 1.0501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8460 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1176 2.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3894 1.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6541 -1.5837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6255 2.3411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2132 -0.8725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2223 0.9716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6542 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0073 3.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1928 2.9103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6629 2.6984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2919 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1435 2.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7292 3.1178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6255 3.4837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7068 1.9544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2081 -1.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4616 -1.5154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1372 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5157 -2.5695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2622 -2.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5867 -1.9940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8427 -0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3251 -1.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4174 -2.8280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5465 4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0687 -3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1412 -4.0563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
6 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
22 26 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
20 32 1 0 0 0 0
23 40 1 0 0 0 0
41 42 1 0 0 0 0
34 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.5530 0.5980
S SKP 5
ID FL5FFAGL0002
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES C(C(C1O)OC(OC(C2=O)=C(c(c5)ccc(O)c5)Oc(c(OC(C4O)OCC(C4O)O)3)c2c(O)cc3O)C(O)C(O)1)O
M END