Mol:FL5FECNS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 26 28 0 0 0 0 0 0 0 0999 V2000 | + | 26 28 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9748 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9748 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9748 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9748 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4744 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4744 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9739 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9739 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9739 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9739 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4744 0.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4744 0.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4735 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4735 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0270 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0270 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0270 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0270 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4735 0.4074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4735 0.4074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5271 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5271 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0575 0.1010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0575 0.1010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5878 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5878 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5878 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5878 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0575 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0575 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5271 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5271 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4744 -1.3258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4744 -1.3258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4735 -1.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4735 -1.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1177 1.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1177 1.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4749 -0.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4749 -0.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3320 0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3320 0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8320 1.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8320 1.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8930 -0.9594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8930 -0.9594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7590 -1.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7590 -1.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4538 -0.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4538 -0.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4538 -0.0928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4538 -0.0928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 7 18 2 0 0 0 0 | + | 7 18 2 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 2 20 1 0 0 0 0 | + | 2 20 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 8 23 1 0 0 0 0 | + | 8 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 13 25 1 0 0 0 0 | + | 13 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 25 26 | + | M SAL 3 2 25 26 |
| − | M SBL 3 1 27 | + | M SBL 3 1 27 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 27 1.7605 0.201 | + | M SVB 3 27 1.7605 0.201 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 23 24 | + | M SAL 2 2 23 24 |
| − | M SBL 2 1 25 | + | M SBL 2 1 25 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 25 0.1699 -0.6656 | + | M SVB 2 25 0.1699 -0.6656 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 21 22 | + | M SAL 1 2 21 22 |
| − | M SBL 1 1 23 | + | M SBL 1 1 23 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 23 -2.332 0.7372 | + | M SVB 1 23 -2.332 0.7372 |
| − | S SKP 8 | + | S SKP 8 |
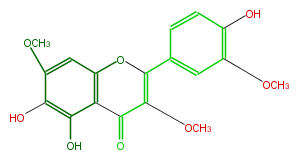
| − | ID FL5FECNS0001 | + | ID FL5FECNS0001 |
| − | KNApSAcK_ID C00001031 | + | KNApSAcK_ID C00001031 |
| − | NAME Chrysosplenol C | + | NAME Chrysosplenol C |
| − | CAS_RN 23370-16-3 | + | CAS_RN 23370-16-3 |
| − | FORMULA C18H16O8 | + | FORMULA C18H16O8 |
| − | EXACTMASS 360.08451748799996 | + | EXACTMASS 360.08451748799996 |
| − | AVERAGEMASS 360.31484 | + | AVERAGEMASS 360.31484 |
| − | SMILES c(c3O)(OC)cc(O1)c(c3O)C(C(=C1c(c2)cc(OC)c(O)c2)OC)=O | + | SMILES c(c3O)(OC)cc(O1)c(c3O)C(C(=C1c(c2)cc(OC)c(O)c2)OC)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
26 28 0 0 0 0 0 0 0 0999 V2000
-1.9748 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9748 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4744 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9739 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9739 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4744 0.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4735 -0.7483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0270 -0.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0270 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4735 0.4074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5271 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0575 0.1010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5878 0.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5878 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0575 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5271 1.0196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4744 -1.3258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4735 -1.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1177 1.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4749 -0.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3320 0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8320 1.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8930 -0.9594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7590 -1.4594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4538 -0.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4538 -0.0928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
3 17 1 0 0 0 0
7 18 2 0 0 0 0
14 19 1 0 0 0 0
2 20 1 0 0 0 0
1 21 1 0 0 0 0
21 22 1 0 0 0 0
8 23 1 0 0 0 0
23 24 1 0 0 0 0
13 25 1 0 0 0 0
25 26 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 25 26
M SBL 3 1 27
M SMT 3 OCH3
M SVB 3 27 1.7605 0.201
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 23 24
M SBL 2 1 25
M SMT 2 OCH3
M SVB 2 25 0.1699 -0.6656
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 21 22
M SBL 1 1 23
M SMT 1 OCH3
M SVB 1 23 -2.332 0.7372
S SKP 8
ID FL5FECNS0001
KNApSAcK_ID C00001031
NAME Chrysosplenol C
CAS_RN 23370-16-3
FORMULA C18H16O8
EXACTMASS 360.08451748799996
AVERAGEMASS 360.31484
SMILES c(c3O)(OC)cc(O1)c(c3O)C(C(=C1c(c2)cc(OC)c(O)c2)OC)=O
M END