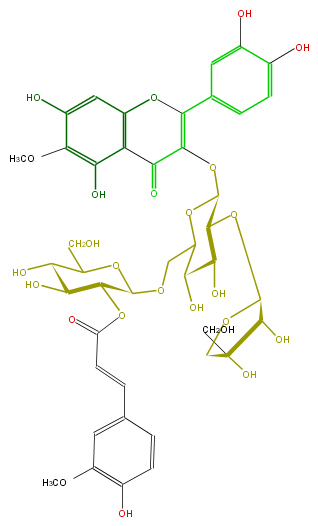
Mol:FL5FECGS0059
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 68 74 0 0 0 0 0 0 0 0999 V2000 | + | 68 74 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.1460 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1460 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4316 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4316 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4316 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4316 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1460 5.3173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1460 5.3173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8605 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8605 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8605 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8605 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7171 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7171 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0026 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0026 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7118 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7118 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7118 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7118 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0026 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0026 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7171 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7171 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4263 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4263 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1408 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1408 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1408 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1408 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4263 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4263 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0026 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0026 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8552 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8552 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4691 2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4691 2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4263 1.6914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4263 1.6914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5750 5.3173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5750 5.3173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1460 6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1460 6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7730 -0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7730 -0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5075 0.0583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5075 0.0583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3387 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3387 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6052 1.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6052 1.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8706 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8706 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0393 0.4634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0393 0.4634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3760 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3760 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1904 -0.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1904 -0.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9724 -1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9724 -1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5075 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5075 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9848 1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9848 1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8181 -2.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8181 -2.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2603 -2.7098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2603 -2.7098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0771 -1.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0771 -1.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6389 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6389 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3053 -2.2795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3053 -2.2795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6417 -1.4171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6417 -1.4171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7868 -1.4105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7868 -1.4105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5136 -0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5136 -0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1010 -0.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1010 -0.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3027 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3027 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5092 -0.7444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5092 -0.7444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9217 -0.0296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9217 -0.0296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7201 -0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7201 -0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2185 -0.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2185 -0.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8749 -0.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8749 -0.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7728 -1.2607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7728 -1.2607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3797 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3797 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0012 -1.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0012 -1.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4030 -2.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4030 -2.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7171 -3.0056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7171 -3.0056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7249 -3.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7249 -3.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4507 -4.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4507 -4.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4741 -5.0195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4741 -5.0195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7716 -5.4520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7716 -5.4520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0460 -5.0600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0460 -5.0600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0226 -4.2353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0226 -4.2353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7716 -6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7716 -6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1402 0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1402 0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0439 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0439 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8580 2.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8580 2.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5750 2.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5750 2.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0670 -5.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0670 -5.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0876 -6.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0876 -6.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1663 -1.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1663 -1.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6175 -1.7724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6175 -1.7724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 39 34 1 1 0 0 0 | + | 39 34 1 1 0 0 0 |
| − | 38 34 1 1 0 0 0 | + | 38 34 1 1 0 0 0 |
| − | 37 39 1 1 0 0 0 | + | 37 39 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 39 36 1 0 0 0 0 | + | 39 36 1 0 0 0 0 |
| − | 40 37 1 0 0 0 0 | + | 40 37 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 44 30 1 0 0 0 0 | + | 44 30 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 46 61 1 0 0 0 0 | + | 46 61 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 15 63 1 0 0 0 0 | + | 15 63 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 56 65 1 0 0 0 0 | + | 56 65 1 0 0 0 0 |
| − | 67 68 1 0 0 0 0 | + | 67 68 1 0 0 0 0 |
| − | 34 67 1 0 0 0 0 | + | 34 67 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 68 | + | M SBL 1 1 68 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 68 0.4201 -0.7277 | + | M SBV 1 68 0.4201 -0.7277 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 63 64 | + | M SAL 2 2 63 64 |
| − | M SBL 2 1 70 | + | M SBL 2 1 70 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 70 0.7172 0.2609 | + | M SBV 2 70 0.7172 0.2609 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 65 66 | + | M SAL 3 2 65 66 |
| − | M SBL 3 1 72 | + | M SBL 3 1 72 |
| − | M SMT 3 ^ OCH3 | + | M SMT 3 ^ OCH3 |
| − | M SBV 3 72 0.5929 0.3423 | + | M SBV 3 72 0.5929 0.3423 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 67 68 | + | M SAL 4 2 67 68 |
| − | M SBL 4 1 74 | + | M SBL 4 1 74 |
| − | M SMT 4 ^ CH2OH | + | M SMT 4 ^ CH2OH |
| − | M SBV 4 74 0.6518 -0.6518 | + | M SBV 4 74 0.6518 -0.6518 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0059 | + | ID FL5FECGS0059 |
| − | FORMULA C43H48O25 | + | FORMULA C43H48O25 |
| − | EXACTMASS 964.2484670859999 | + | EXACTMASS 964.2484670859999 |
| − | AVERAGEMASS 964.82622 | + | AVERAGEMASS 964.82622 |
| − | SMILES C(OCC(O3)C(O)C(O)C(OC(C7O)OCC(O)7CO)C3OC(=C5c(c6)cc(O)c(c6)O)C(c(c4O)c(O5)cc(O)c(OC)4)=O)(O2)C(C(O)C(C2CO)O)OC(C=Cc(c1)cc(OC)c(O)c1)=O | + | SMILES C(OCC(O3)C(O)C(O)C(OC(C7O)OCC(O)7CO)C3OC(=C5c(c6)cc(O)c(c6)O)C(c(c4O)c(O5)cc(O)c(OC)4)=O)(O2)C(C(O)C(C2CO)O)OC(C=Cc(c1)cc(OC)c(O)c1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
68 74 0 0 0 0 0 0 0 0999 V2000
2.1460 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4316 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4316 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1460 5.3173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8605 4.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8605 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7171 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0026 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7118 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7118 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0026 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7171 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4263 4.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1408 3.6673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1408 2.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4263 2.4298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0026 1.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8552 4.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4691 2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4263 1.6914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5750 5.3173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1460 6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7730 -0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5075 0.0583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3387 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6052 1.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8706 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0393 0.4634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3760 0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1904 -0.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9724 -1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5075 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9848 1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8181 -2.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2603 -2.7098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0771 -1.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6389 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3053 -2.2795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6417 -1.4171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7868 -1.4105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5136 -0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1010 -0.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3027 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5092 -0.7444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9217 -0.0296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7201 -0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2185 -0.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8749 -0.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7728 -1.2607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3797 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0012 -1.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4030 -2.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7171 -3.0056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7249 -3.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4507 -4.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4741 -5.0195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7716 -5.4520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0460 -5.0600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0226 -4.2353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7716 -6.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1402 0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0439 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8580 2.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5750 2.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0670 -5.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0876 -6.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1663 -1.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6175 -1.7724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
39 34 1 1 0 0 0
38 34 1 1 0 0 0
37 39 1 1 0 0 0
34 35 1 0 0 0 0
39 36 1 0 0 0 0
40 37 1 0 0 0 0
38 40 1 0 0 0 0
33 37 1 0 0 0 0
26 19 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
50 51 2 0 0 0 0
50 52 1 0 0 0 0
52 53 2 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 54 1 0 0 0 0
57 60 1 0 0 0 0
49 50 1 0 0 0 0
44 30 1 0 0 0 0
61 62 1 0 0 0 0
46 61 1 0 0 0 0
63 64 1 0 0 0 0
15 63 1 0 0 0 0
65 66 1 0 0 0 0
56 65 1 0 0 0 0
67 68 1 0 0 0 0
34 67 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 68
M SMT 1 ^ CH2OH
M SBV 1 68 0.4201 -0.7277
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 63 64
M SBL 2 1 70
M SMT 2 ^ OCH3
M SBV 2 70 0.7172 0.2609
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 65 66
M SBL 3 1 72
M SMT 3 ^ OCH3
M SBV 3 72 0.5929 0.3423
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 67 68
M SBL 4 1 74
M SMT 4 ^ CH2OH
M SBV 4 74 0.6518 -0.6518
S SKP 5
ID FL5FECGS0059
FORMULA C43H48O25
EXACTMASS 964.2484670859999
AVERAGEMASS 964.82622
SMILES C(OCC(O3)C(O)C(O)C(OC(C7O)OCC(O)7CO)C3OC(=C5c(c6)cc(O)c(c6)O)C(c(c4O)c(O5)cc(O)c(OC)4)=O)(O2)C(C(O)C(C2CO)O)OC(C=Cc(c1)cc(OC)c(O)c1)=O
M END