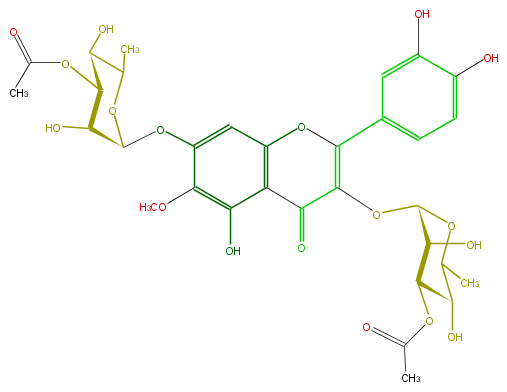
Mol:FL5FECGS0045
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 54 0 0 0 0 0 0 0 0999 V2000 | + | 50 54 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1190 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1190 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1190 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1190 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3100 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3100 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3100 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3100 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 1.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 1.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0244 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0244 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7389 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7389 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7389 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7389 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0244 1.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0244 1.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0244 -1.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0244 -1.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6385 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6385 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3666 1.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3666 1.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0948 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0948 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0948 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0948 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3666 2.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3666 2.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6385 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6385 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 -1.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 -1.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7518 2.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7518 2.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8005 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8005 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5308 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5308 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3426 -1.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3426 -1.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0302 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0302 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8120 -1.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8120 -1.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0302 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0302 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3426 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3426 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5607 -0.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5607 -0.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4038 -0.9763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4038 -0.9763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5824 -2.5080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5824 -2.5080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0316 -2.8326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0316 -2.8326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2860 -1.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2860 -1.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2121 1.3333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2121 1.3333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5376 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5376 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7515 1.6927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7515 1.6927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5376 2.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5376 2.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2121 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2121 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9981 2.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9981 2.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9453 3.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9453 3.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5142 2.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5142 2.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6873 2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6873 2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8631 1.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8631 1.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3666 3.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3666 3.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4079 2.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4079 2.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7347 3.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7347 3.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7518 2.0458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7518 2.0458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0814 -3.0062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0814 -3.0062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3375 -2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3375 -2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0921 -3.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0921 -3.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7595 -0.2322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7595 -0.2322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8419 -0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8419 -0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 46 29 1 0 0 0 0 | + | 46 29 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 2 49 1 0 0 0 0 | + | 2 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 54 0.6405 0.3698 | + | M SBV 1 54 0.6405 0.3698 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0045 | + | ID FL5FECGS0045 |
| − | FORMULA C32H36O18 | + | FORMULA C32H36O18 |
| − | EXACTMASS 708.190164348 | + | EXACTMASS 708.190164348 |
| − | AVERAGEMASS 708.61744 | + | AVERAGEMASS 708.61744 |
| − | SMILES OC(C1OC(=C4c(c5)cc(O)c(O)c5)C(=O)c(c(O)3)c(O4)cc(c3OC)OC(C2O)OC(C)C(C2OC(C)=O)O)C(C(O)C(C)O1)OC(C)=O | + | SMILES OC(C1OC(=C4c(c5)cc(O)c(O)c5)C(=O)c(c(O)3)c(O4)cc(c3OC)OC(C2O)OC(C)C(C2OC(C)=O)O)C(C(O)C(C)O1)OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 54 0 0 0 0 0 0 0 0999 V2000
-1.1190 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1190 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3100 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3100 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 1.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0244 -0.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7389 0.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7389 0.9625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0244 1.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0244 -1.0404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6385 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3666 1.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0948 1.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0948 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3666 2.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6385 2.3746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 -1.0996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7518 2.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8005 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5308 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3426 -1.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0302 -2.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8120 -1.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0302 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3426 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5607 -0.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4038 -0.9763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5824 -2.5080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0316 -2.8326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2860 -1.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2121 1.3333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5376 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7515 1.6927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5376 2.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2121 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9981 2.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9453 3.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5142 2.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6873 2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8631 1.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3666 3.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4079 2.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7347 3.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7518 2.0458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0814 -3.0062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3375 -2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0921 -3.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7595 -0.2322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8419 -0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
20 33 1 0 0 0 0
16 42 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
40 43 1 0 0 0 0
46 47 2 0 0 0 0
46 48 1 0 0 0 0
46 29 1 0 0 0 0
49 50 1 0 0 0 0
2 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 54
M SMT 1 ^OCH3
M SBV 1 54 0.6405 0.3698
S SKP 5
ID FL5FECGS0045
FORMULA C32H36O18
EXACTMASS 708.190164348
AVERAGEMASS 708.61744
SMILES OC(C1OC(=C4c(c5)cc(O)c(O)c5)C(=O)c(c(O)3)c(O4)cc(c3OC)OC(C2O)OC(C)C(C2OC(C)=O)O)C(C(O)C(C)O1)OC(C)=O
M END