Mol:FL5FCJGA0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1567 -5.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1567 -5.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4759 -5.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4759 -5.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6956 -4.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6956 -4.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2827 -3.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2827 -3.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3499 -4.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3499 -4.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5696 -4.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5696 -4.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5024 -3.3776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5024 -3.3776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0895 -2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0895 -2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5431 -2.9970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5431 -2.9970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7628 -3.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7628 -3.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9956 -3.2905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9956 -3.2905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9559 -2.5051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9559 -2.5051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7319 -1.8900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7319 -1.8900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1528 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1528 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7975 -1.5021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7975 -1.5021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0214 -2.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0214 -2.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6006 -2.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6006 -2.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5189 -2.1517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5189 -2.1517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9696 -0.8011 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9696 -0.8011 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5820 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5820 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3274 -1.2595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3274 -1.2595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0774 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0774 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4651 -0.8011 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4651 -0.8011 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7196 -1.0141 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7196 -1.0141 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2425 -0.9960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2425 -0.9960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9884 -0.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9884 -0.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4651 -0.1376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4651 -0.1376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3278 -4.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3278 -4.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6659 -2.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6659 -2.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9289 -0.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9289 -0.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5919 -1.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5919 -1.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3064 -1.8577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3064 -1.8577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7040 -5.6477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7040 -5.6477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4700 -6.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4700 -6.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2182 -1.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2182 -1.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9360 -0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9360 -0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 16 29 1 0 0 0 0 | + | 16 29 1 0 0 0 0 |
| − | 14 30 1 0 0 0 0 | + | 14 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 1 33 1 0 0 0 0 | + | 1 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 15 35 1 0 0 0 0 | + | 15 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 31 32 | + | M SAL 3 2 31 32 |
| − | M SBL 3 1 34 | + | M SBL 3 1 34 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 34 2.5919 -1.4452 | + | M SVB 3 34 2.5919 -1.4452 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 35 36 | + | M SAL 2 2 35 36 |
| − | M SBL 2 1 38 | + | M SBL 2 1 38 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 38 1.5329 1.2031 | + | M SVB 2 38 1.5329 1.2031 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 33 34 | + | M SAL 1 2 33 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 36 -3.3064 0.5189 | + | M SVB 1 36 -3.3064 0.5189 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCJGA0001 | + | ID FL5FCJGA0001 |
| − | KNApSAcK_ID C00005774 | + | KNApSAcK_ID C00005774 |
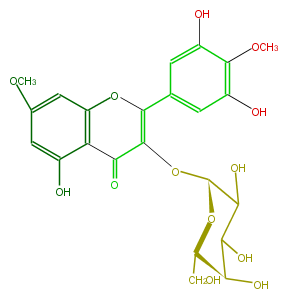
| − | NAME Myricetin 7,4'-dimethyl ether 3-galactoside | + | NAME Myricetin 7,4'-dimethyl ether 3-galactoside |
| − | CAS_RN 86828-99-1 | + | CAS_RN 86828-99-1 |
| − | FORMULA C23H24O13 | + | FORMULA C23H24O13 |
| − | EXACTMASS 508.121690854 | + | EXACTMASS 508.121690854 |
| − | AVERAGEMASS 508.42886 | + | AVERAGEMASS 508.42886 |
| − | SMILES O([C@H](O4)C(O)C([C@H]([C@@H](CO)4)O)O)C(C(=O)2)=C(c(c3)cc(c(OC)c(O)3)O)Oc(c21)cc(cc1O)OC | + | SMILES O([C@H](O4)C(O)C([C@H]([C@@H](CO)4)O)O)C(C(=O)2)=C(c(c3)cc(c(OC)c(O)3)O)Oc(c21)cc(cc1O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-0.1567 -5.1884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4759 -5.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6956 -4.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2827 -3.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3499 -4.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5696 -4.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5024 -3.3776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0895 -2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5431 -2.9970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7628 -3.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9956 -3.2905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9559 -2.5051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7319 -1.8900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1528 -1.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7975 -1.5021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0214 -2.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6006 -2.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5189 -2.1517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9696 -0.8011 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5820 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3274 -1.2595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0774 -1.4726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4651 -0.8011 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7196 -1.0141 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2425 -0.9960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9884 -0.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4651 -0.1376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3278 -4.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6659 -2.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9289 -0.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5919 -1.4452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3064 -1.8577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7040 -5.6477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4700 -6.2906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2182 -1.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9360 -0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
3 28 1 0 0 0 0
16 29 1 0 0 0 0
14 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
1 33 1 0 0 0 0
33 34 1 0 0 0 0
15 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 31 32
M SBL 3 1 34
M SMT 3 CH2OH
M SVB 3 34 2.5919 -1.4452
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 35 36
M SBL 2 1 38
M SMT 2 OCH3
M SVB 2 38 1.5329 1.2031
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 33 34
M SBL 1 1 36
M SMT 1 OCH3
M SVB 1 36 -3.3064 0.5189
S SKP 8
ID FL5FCJGA0001
KNApSAcK_ID C00005774
NAME Myricetin 7,4'-dimethyl ether 3-galactoside
CAS_RN 86828-99-1
FORMULA C23H24O13
EXACTMASS 508.121690854
AVERAGEMASS 508.42886
SMILES O([C@H](O4)C(O)C([C@H]([C@@H](CO)4)O)O)C(C(=O)2)=C(c(c3)cc(c(OC)c(O)3)O)Oc(c21)cc(cc1O)OC
M END