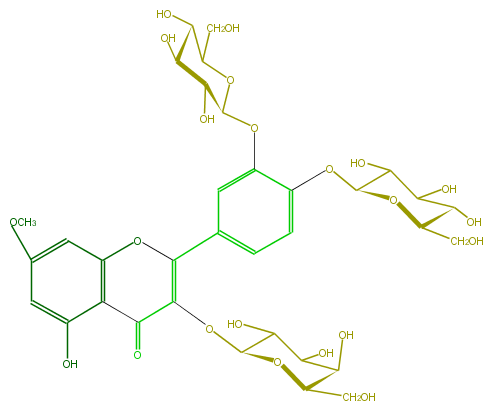
Mol:FL5FCCGA0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.1675 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1675 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1675 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1675 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4089 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4089 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6503 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6503 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6503 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6503 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4089 -0.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4089 -0.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8917 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8917 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1331 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1331 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1331 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1331 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8917 -0.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8917 -0.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8917 -2.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8917 -2.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1779 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1779 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5951 -0.4728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5951 -0.4728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3683 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3683 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3683 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3683 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5951 1.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5951 1.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1779 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1779 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4089 -2.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4089 -2.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1958 1.3441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1958 1.3441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3650 -2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3650 -2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5951 2.2053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5951 2.2053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7428 4.3982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7428 4.3982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0683 3.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0683 3.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4584 3.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4584 3.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1002 2.5312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1002 2.5312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0870 3.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0870 3.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5234 3.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5234 3.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2675 4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2675 4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3129 4.1348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3129 4.1348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4841 2.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4841 2.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4850 1.2852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4850 1.2852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7744 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7744 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5703 0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5703 0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1448 0.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1448 0.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8554 0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8554 0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0596 0.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0596 0.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8895 1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8895 1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6810 0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6810 0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2124 0.2130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2124 0.2130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0136 -2.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0136 -2.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3031 -2.5881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3031 -2.5881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0991 -2.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0991 -2.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6736 -3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6736 -3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3842 -2.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3842 -2.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5882 -2.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5882 -2.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2517 -1.9737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2517 -1.9737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0448 -2.5897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0448 -2.5897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4673 -2.2049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4673 -2.2049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6547 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6547 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3367 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3367 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8612 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8612 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3367 -1.2275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3367 -1.2275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3488 4.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3488 4.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7378 4.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7378 4.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5611 -3.5295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5611 -3.5295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5611 -4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5611 -4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 19 1 0 0 0 0 | + | 32 19 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 41 20 1 0 0 0 0 | + | 41 20 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 1 49 1 0 0 0 0 | + | 1 49 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 34 51 1 0 0 0 0 | + | 34 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 27 53 1 0 0 0 0 | + | 27 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 43 55 1 0 0 0 0 | + | 43 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 55 0.4872 -0.8437 | + | M SBV 1 55 0.4872 -0.8437 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 57 -0.7164 0.2916 | + | M SBV 2 57 -0.7164 0.2916 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 53 54 | + | M SAL 3 2 53 54 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 59 -0.1745 -0.7299 | + | M SBV 3 59 -0.1745 -0.7299 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 55 56 | + | M SAL 4 2 55 56 |
| − | M SBL 4 1 61 | + | M SBL 4 1 61 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SBV 4 61 -0.8875 0.1503 | + | M SBV 4 61 -0.8875 0.1503 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCCGA0007 | + | ID FL5FCCGA0007 |
| − | FORMULA C34H42O22 | + | FORMULA C34H42O22 |
| − | EXACTMASS 802.216773028 | + | EXACTMASS 802.216773028 |
| − | AVERAGEMASS 802.68408 | + | AVERAGEMASS 802.68408 |
| − | SMILES C(O)(C(O)6)C(O)C(OC6CO)Oc(c4)c(OC(O5)C(O)C(C(O)C(CO)5)O)cc(c4)C(O3)=C(C(=O)c(c32)c(O)cc(c2)OC)OC(C(O)1)OC(CO)C(O)C1O | + | SMILES C(O)(C(O)6)C(O)C(OC6CO)Oc(c4)c(OC(O5)C(O)C(C(O)C(CO)5)O)cc(c4)C(O3)=C(C(=O)c(c32)c(O)cc(c2)OC)OC(C(O)1)OC(CO)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-4.1675 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1675 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4089 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6503 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6503 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4089 -0.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8917 -1.9469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1331 -1.5089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1331 -0.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8917 -0.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8917 -2.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1779 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5951 -0.4728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3683 -0.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3683 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5951 1.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1779 0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4089 -2.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1958 1.3441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3650 -2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5951 2.2053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7428 4.3982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0683 3.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4584 3.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1002 2.5312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0870 3.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5234 3.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2675 4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3129 4.1348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4841 2.4022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4850 1.2852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7744 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5703 0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1448 0.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8554 0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0596 0.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8895 1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6810 0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2124 0.2130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0136 -2.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3031 -2.5881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0991 -2.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6736 -3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3842 -2.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5882 -2.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2517 -1.9737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0448 -2.5897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4673 -2.2049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6547 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3367 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8612 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3367 -1.2275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3488 4.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7378 4.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5611 -3.5295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5611 -4.6544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 19 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
41 20 1 0 0 0 0
49 50 1 0 0 0 0
1 49 1 0 0 0 0
51 52 1 0 0 0 0
34 51 1 0 0 0 0
53 54 1 0 0 0 0
27 53 1 0 0 0 0
55 56 1 0 0 0 0
43 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 55
M SMT 1 ^ OCH3
M SBV 1 55 0.4872 -0.8437
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 57
M SMT 2 CH2OH
M SBV 2 57 -0.7164 0.2916
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 53 54
M SBL 3 1 59
M SMT 3 CH2OH
M SBV 3 59 -0.1745 -0.7299
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 55 56
M SBL 4 1 61
M SMT 4 CH2OH
M SBV 4 61 -0.8875 0.1503
S SKP 5
ID FL5FCCGA0007
FORMULA C34H42O22
EXACTMASS 802.216773028
AVERAGEMASS 802.68408
SMILES C(O)(C(O)6)C(O)C(OC6CO)Oc(c4)c(OC(O5)C(O)C(C(O)C(CO)5)O)cc(c4)C(O3)=C(C(=O)c(c32)c(O)cc(c2)OC)OC(C(O)1)OC(CO)C(O)C1O
M END