Mol:FL5FCCGA0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6554 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6554 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6554 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6554 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9409 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9409 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2265 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2265 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2265 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2265 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9409 -0.6982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9409 -0.6982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5120 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5120 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2025 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2025 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2025 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2025 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5120 -0.6982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5120 -0.6982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5120 -2.9913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5120 -2.9913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1021 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1021 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8302 -0.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8302 -0.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5584 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5584 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5584 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5584 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8302 0.7219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8302 0.7219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1021 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1021 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9409 -3.1728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9409 -3.1728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3377 0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3377 0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0510 -2.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0510 -2.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8302 1.5625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8302 1.5625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9964 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9964 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6101 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6101 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3531 -2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3531 -2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1004 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1004 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4869 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4869 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7439 -2.2320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7439 -2.2320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3834 -2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3834 -2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0547 -1.8034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0547 -1.8034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4626 -1.2792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4626 -1.2792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1999 3.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1999 3.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5282 2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5282 2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3357 2.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3357 2.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0435 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0435 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6681 3.0225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6681 3.0225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9378 2.8561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9378 2.8561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5991 3.0807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5991 3.0807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2850 2.6785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2850 2.6785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6554 3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6554 3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6135 3.4334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6135 3.4334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8983 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8983 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8983 -3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8983 -3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2561 -0.7638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2561 -0.7638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8983 0.3484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8983 0.3484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 21 34 1 0 0 0 0 | + | 21 34 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 1 43 1 0 0 0 0 | + | 1 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.7978 -0.1220 | + | M SBV 1 46 -0.7978 -0.1220 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 48 0.6007 -0.3468 | + | M SBV 2 48 0.6007 -0.3468 |
| − | S SKP 5 | + | S SKP 5 |
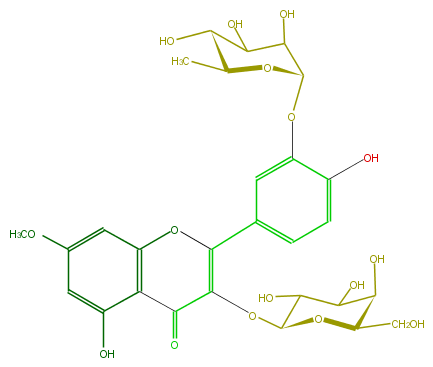
| − | ID FL5FCCGA0005 | + | ID FL5FCCGA0005 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C1OC(=C3c(c4)cc(OC(O5)C(C(C(C(C)5)O)O)O)c(c4)O)C(c(c2O)c(O3)cc(c2)OC)=O)C(C(O)C(O1)CO)O | + | SMILES OC(C1OC(=C3c(c4)cc(OC(O5)C(C(C(C(C)5)O)O)O)c(c4)O)C(c(c2O)c(O3)cc(c2)OC)=O)C(C(O)C(O1)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.6554 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6554 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9409 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2265 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2265 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9409 -0.6982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5120 -2.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2025 -1.9356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2025 -1.1107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5120 -0.6982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5120 -2.9913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1021 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8302 -0.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5584 -0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5584 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8302 0.7219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1021 0.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9409 -3.1728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3377 0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0510 -2.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8302 1.5625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9964 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6101 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3531 -2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1004 -2.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4869 -2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7439 -2.2320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3834 -2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0547 -1.8034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4626 -1.2792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1999 3.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5282 2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3357 2.5536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0435 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6681 3.0225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9378 2.8561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5991 3.0807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2850 2.6785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6554 3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6135 3.4334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8983 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8983 -3.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2561 -0.7638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8983 0.3484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
35 39 1 0 0 0 0
36 40 1 0 0 0 0
21 34 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
43 44 1 0 0 0 0
1 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.7978 -0.1220
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 ^ OCH3
M SBV 2 48 0.6007 -0.3468
S SKP 5
ID FL5FCCGA0005
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C1OC(=C3c(c4)cc(OC(O5)C(C(C(C(C)5)O)O)O)c(c4)O)C(c(c2O)c(O3)cc(c2)OC)=O)C(C(O)C(O1)CO)O
M END