Mol:FL5FBAGA0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8477 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8477 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8477 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8477 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2914 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2914 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7351 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7351 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7351 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7351 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2914 0.5491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2914 0.5491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1788 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1788 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6225 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6225 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6225 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6225 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1788 0.5491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1788 0.5491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1788 -1.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1788 -1.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0664 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0664 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5006 0.2216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5006 0.2216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0675 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0675 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0675 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0675 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5006 1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5006 1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0664 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0664 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4038 0.5490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4038 0.5490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0256 -0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0256 -0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0710 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0710 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6834 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6834 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4288 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4288 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1788 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1788 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5665 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5665 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8210 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8210 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3439 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3439 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0856 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0856 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3305 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3305 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6343 1.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6343 1.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7278 -0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7278 -0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0134 -1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0134 -1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6893 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6893 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4038 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4038 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 33 -5.6931 5.5330 | + | M SBV 1 33 -5.6931 5.5330 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 35 -4.7462 5.7198 | + | M SBV 2 35 -4.7462 5.7198 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FBAGA0001 | + | ID FL5FBAGA0001 |
| − | KNApSAcK_ID C00005267 | + | KNApSAcK_ID C00005267 |
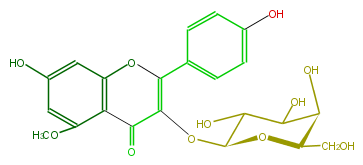
| − | NAME Kaempferol 5-methyl ether 3-galactoside | + | NAME Kaempferol 5-methyl ether 3-galactoside |
| − | CAS_RN 28454-84-4 | + | CAS_RN 28454-84-4 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c(OC)3)c2cc(c3)O)OC(O1)C(O)C(C(C(CO)1)O)O)O | + | SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c(OC)3)c2cc(c3)O)OC(O1)C(O)C(C(C(CO)1)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-2.8477 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8477 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2914 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7351 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7351 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2914 0.5491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1788 -0.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6225 -0.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6225 0.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1788 0.5491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1788 -1.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0664 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5006 0.2216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0675 0.5490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0675 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5006 1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0664 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4038 0.5490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0256 -0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0710 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6834 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4288 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1788 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5665 -0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8210 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3439 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0856 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3305 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6343 1.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7278 -0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0134 -1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6893 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4038 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
23 22 1 1 0 0 0
15 29 1 0 0 0 0
3 30 1 0 0 0 0
30 31 1 0 0 0 0
23 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SBV 1 33 -5.6931 5.5330
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 CH2OH
M SBV 2 35 -4.7462 5.7198
S SKP 8
ID FL5FBAGA0001
KNApSAcK_ID C00005267
NAME Kaempferol 5-methyl ether 3-galactoside
CAS_RN 28454-84-4
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c(OC)3)c2cc(c3)O)OC(O1)C(O)C(C(C(CO)1)O)O)O
M END