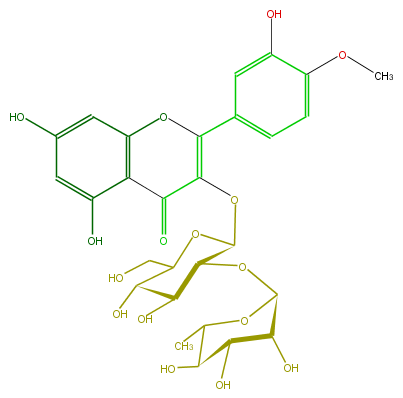
Mol:FL5FAEGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.4295 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4295 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7151 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7151 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7151 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7151 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4295 2.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4295 2.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1439 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1439 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1439 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1439 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0006 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0006 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7139 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7139 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4283 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4283 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4283 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4283 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7139 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7139 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0006 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0006 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1427 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1427 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8572 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8572 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8572 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8572 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1427 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1427 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7139 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7139 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5717 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5717 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7151 0.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7151 0.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1427 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1427 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8584 2.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8584 2.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4295 3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4295 3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5717 2.4760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5717 2.4760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2717 -1.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2717 -1.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4868 -1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4868 -1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0339 -1.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0339 -1.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7074 -0.9435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7074 -0.9435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0776 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0776 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5306 -1.3787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5306 -1.3787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9968 -1.2357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9968 -1.2357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5992 -1.5589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5992 -1.5589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6564 -2.2810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6564 -2.2810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1474 -2.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1474 -2.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8668 -1.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8668 -1.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0889 -2.5355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0889 -2.5355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0254 -3.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0254 -3.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6249 -2.8449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6249 -2.8449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4434 -2.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4434 -2.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5579 -1.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5579 -1.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9073 -2.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9073 -2.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7735 -3.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7735 -3.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4166 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4166 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5609 -3.3851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5609 -3.3851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4169 -2.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4169 -2.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 35 44 1 0 0 0 0 | + | 35 44 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAEGS0005 | + | ID FL5FAEGS0005 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C1O)C(OC(C(OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c(O)3)2)C(O)C(O)C(CO)O2)OC(C)C1O | + | SMILES OC(C1O)C(OC(C(OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c(O)3)2)C(O)C(O)C(CO)O2)OC(C)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
1.4295 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7151 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7151 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4295 2.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1439 2.4753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1439 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0006 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7139 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4283 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4283 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7139 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0006 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1427 1.6503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8572 1.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8572 0.4129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1427 0.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7139 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5717 1.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7151 0.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1427 -0.8246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8584 2.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4295 3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5717 2.4760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2717 -1.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4868 -1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0339 -1.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7074 -0.9435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0776 -0.6889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5306 -1.3787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9968 -1.2357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5992 -1.5589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6564 -2.2810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1474 -2.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8668 -1.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0889 -2.5355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0254 -3.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6249 -2.8449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4434 -2.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5579 -1.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9073 -2.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7735 -3.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4166 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5609 -3.3851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4169 -2.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
21 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
37 42 1 0 0 0 0
36 43 1 0 0 0 0
35 44 1 0 0 0 0
39 34 1 0 0 0 0
27 19 1 0 0 0 0
S SKP 5
ID FL5FAEGS0005
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C1O)C(OC(C(OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c(O)3)2)C(O)C(O)C(CO)O2)OC(C)C1O
M END