Mol:FL5FAEGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2997 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2997 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2997 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2997 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7434 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7434 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1871 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1871 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1871 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1871 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7434 0.8175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7434 0.8175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6308 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6308 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0745 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0745 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0745 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0745 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6308 0.8175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6308 0.8175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6308 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6308 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4816 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4816 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0486 0.4901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0486 0.4901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6155 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6155 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6155 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6155 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0486 1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0486 1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4816 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4816 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8558 0.8174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8558 0.8174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7434 -1.1093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7434 -1.1093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4816 -0.4671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4816 -0.4671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0486 2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0486 2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2940 -1.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2940 -1.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9222 -1.8883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9222 -1.8883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7229 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7229 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9222 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9222 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2940 -0.1263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2940 -0.1263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4933 -0.8238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4933 -0.8238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2636 -0.8238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2636 -0.8238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4554 -2.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4554 -2.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7707 -2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7707 -2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3431 -1.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3431 -1.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1413 1.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1413 1.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8558 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8558 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 15 32 1 0 0 0 0 | + | 15 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 35 -8.0398 6.8964 | + | M SBV 1 35 -8.0398 6.8964 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAEGS0001 | + | ID FL5FAEGS0001 |
| − | KNApSAcK_ID C00005592 | + | KNApSAcK_ID C00005592 |
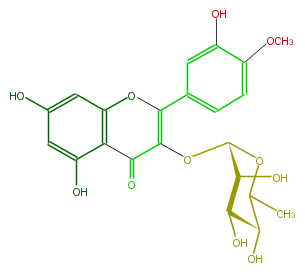
| − | NAME Tamarixetin 3-rhamnoside | + | NAME Tamarixetin 3-rhamnoside |
| − | CAS_RN 87562-18-3 | + | CAS_RN 87562-18-3 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES Oc(c1)c(ccc1C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(O)C(C)2)O)OC | + | SMILES Oc(c1)c(ccc1C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(O)C(C)2)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-2.2997 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2997 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7434 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1871 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1871 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7434 0.8175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6308 -0.4672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0745 -0.1460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0745 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6308 0.8175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6308 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4816 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0486 0.4901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6155 0.8174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6155 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0486 1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4816 1.4721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8558 0.8174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7434 -1.1093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4816 -0.4671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0486 2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2940 -1.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9222 -1.8883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7229 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9222 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2940 -0.1263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4933 -0.8238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2636 -0.8238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4554 -2.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7707 -2.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3431 -1.5489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1413 1.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8558 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
16 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 20 1 0 0 0 0
15 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 OCH3
M SBV 1 35 -8.0398 6.8964
S SKP 8
ID FL5FAEGS0001
KNApSAcK_ID C00005592
NAME Tamarixetin 3-rhamnoside
CAS_RN 87562-18-3
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES Oc(c1)c(ccc1C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(O)C(C)2)O)OC
M END