Mol:FL5FADGL0032
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4789 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4789 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4789 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4789 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9226 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9226 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3663 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3663 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3663 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3663 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9226 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9226 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1900 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1900 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7463 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7463 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7463 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7463 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1900 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1900 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1900 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1900 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4468 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4468 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0137 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0137 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5807 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5807 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5807 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5807 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0137 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0137 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4468 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4468 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9226 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9226 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4467 2.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4467 2.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1434 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1434 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1903 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1903 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4229 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.4229 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1221 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1221 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7006 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7006 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2826 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.2826 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5835 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.5835 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0049 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.0049 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8642 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8642 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0465 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0465 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3072 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3072 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8341 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8341 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6270 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.6270 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.2760 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2760 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.2450 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2450 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.5690 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.5690 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.8573 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8573 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9201 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.9201 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.6270 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6270 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0035 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0035 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8396 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8396 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2483 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2483 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9199 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.9199 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.2911 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2911 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8255 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8255 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3413 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3413 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9665 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9665 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4989 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.4989 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3290 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3290 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9407 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9407 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1318 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1318 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3690 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3690 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7533 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7533 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3257 1.5883 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.3257 1.5883 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.9546 1.0984 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9546 1.0984 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.4201 1.3062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.4201 1.3062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9043 1.3118 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9043 1.3118 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.2791 1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2791 1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7467 1.4398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.7467 1.4398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.9166 1.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9166 1.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3049 0.6372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3049 0.6372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1138 0.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1138 0.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8064 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8064 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0311 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0311 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2971 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2971 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7385 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7385 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4393 1.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4393 1.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7215 2.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7215 2.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 31 51 1 0 0 0 0 | + | 31 51 1 0 0 0 0 |
| − | 51 35 1 0 0 0 0 | + | 51 35 1 0 0 0 0 |
| − | 41 52 1 0 0 0 0 | + | 41 52 1 0 0 0 0 |
| − | 52 45 1 0 0 0 0 | + | 52 45 1 0 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 56 55 1 1 0 0 0 | + | 56 55 1 1 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 20 1 0 0 0 0 | + | 56 20 1 0 0 0 0 |
| − | 47 62 1 0 0 0 0 | + | 47 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 16 64 1 0 0 0 0 | + | 16 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 58 66 1 0 0 0 0 | + | 58 66 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 66 67 | + | M SAL 3 2 66 67 |
| − | M SBL 3 1 72 | + | M SBL 3 1 72 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 72 -4.1452 2.224 | + | M SVB 3 72 -4.1452 2.224 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 62 63 | + | M SAL 2 2 62 63 |
| − | M SBL 2 1 68 | + | M SBL 2 1 68 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 68 1.1005 -1.241 | + | M SVB 2 68 1.1005 -1.241 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 64 65 | + | M SAL 1 2 64 65 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 70 2.2971 3.2436 | + | M SVB 1 70 2.2971 3.2436 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGL0032 | + | ID FL5FADGL0032 |
| − | KNApSAcK_ID C00005589 | + | KNApSAcK_ID C00005589 |
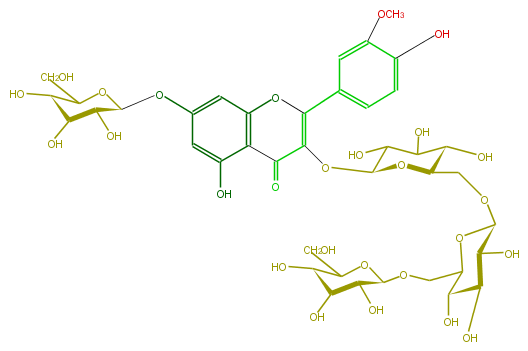
| − | NAME Isorhamnetin 3-gentiotrioside-7-glucoside | + | NAME Isorhamnetin 3-gentiotrioside-7-glucoside |
| − | CAS_RN 84534-27-0 | + | CAS_RN 84534-27-0 |
| − | FORMULA C40H52O27 | + | FORMULA C40H52O27 |
| − | EXACTMASS 964.269596458 | + | EXACTMASS 964.269596458 |
| − | AVERAGEMASS 964.82468 | + | AVERAGEMASS 964.82468 |
| − | SMILES C(C1O)(O)[C@@H](O)[C@@H](CO[C@@H]([C@@H](O)6)OC(CO[C@H](O7)[C@H]([C@H]([C@@H](O)C(CO)7)O)O)[C@H](O)[C@@H]6O)O[C@H]1OC(=C(c(c5)ccc(c(OC)5)O)4)C(c(c3O4)c(cc(c3)O[C@@H]([C@H]2O)OC([C@@H]([C@@H]2O)O)CO)O)=O | + | SMILES C(C1O)(O)[C@@H](O)[C@@H](CO[C@@H]([C@@H](O)6)OC(CO[C@H](O7)[C@H]([C@H]([C@@H](O)C(CO)7)O)O)[C@H](O)[C@@H]6O)O[C@H]1OC(=C(c(c5)ccc(c(OC)5)O)4)C(c(c3O4)c(cc(c3)O[C@@H]([C@H]2O)OC([C@@H]([C@@H]2O)O)CO)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
-1.4789 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4789 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9226 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3663 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3663 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9226 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1900 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7463 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7463 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1900 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1900 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4468 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0137 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5807 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5807 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0137 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4468 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9226 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4467 2.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1434 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1903 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4229 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1221 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7006 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2826 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5835 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0049 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8642 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0465 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3072 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8341 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6270 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.2760 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.2450 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.5690 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.8573 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9201 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.6270 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0035 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8396 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2483 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9199 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.2911 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8255 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3413 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9665 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4989 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3290 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9407 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1318 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3690 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7533 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3257 1.5883 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.9546 1.0984 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.4201 1.3062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9043 1.3118 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.2791 1.6866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7467 1.4398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.9166 1.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3049 0.6372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1138 0.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8064 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0311 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2971 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7385 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4393 1.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7215 2.7574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 21 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
37 41 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
31 51 1 0 0 0 0
51 35 1 0 0 0 0
41 52 1 0 0 0 0
52 45 1 0 0 0 0
53 54 1 1 0 0 0
54 55 1 1 0 0 0
56 55 1 1 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 53 1 0 0 0 0
53 59 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 20 1 0 0 0 0
47 62 1 0 0 0 0
62 63 1 0 0 0 0
16 64 1 0 0 0 0
64 65 1 0 0 0 0
58 66 1 0 0 0 0
66 67 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 66 67
M SBL 3 1 72
M SMT 3 CH2OH
M SVB 3 72 -4.1452 2.224
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 62 63
M SBL 2 1 68
M SMT 2 CH2OH
M SVB 2 68 1.1005 -1.241
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 64 65
M SBL 1 1 70
M SMT 1 OCH3
M SVB 1 70 2.2971 3.2436
S SKP 8
ID FL5FADGL0032
KNApSAcK_ID C00005589
NAME Isorhamnetin 3-gentiotrioside-7-glucoside
CAS_RN 84534-27-0
FORMULA C40H52O27
EXACTMASS 964.269596458
AVERAGEMASS 964.82468
SMILES C(C1O)(O)[C@@H](O)[C@@H](CO[C@@H]([C@@H](O)6)OC(CO[C@H](O7)[C@H]([C@H]([C@@H](O)C(CO)7)O)O)[C@H](O)[C@@H]6O)O[C@H]1OC(=C(c(c5)ccc(c(OC)5)O)4)C(c(c3O4)c(cc(c3)O[C@@H]([C@H]2O)OC([C@@H]([C@@H]2O)O)CO)O)=O
M END