Mol:FL5FACGL0058
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1971 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1971 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1971 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1971 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6408 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6408 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0845 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0845 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0845 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0845 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6408 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6408 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5282 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5282 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9719 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9719 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9719 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9719 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5282 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5282 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5282 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5282 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4158 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4158 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1511 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1511 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7181 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7181 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7181 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7181 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1511 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1511 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4158 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4158 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6408 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6408 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5118 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5118 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6921 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6921 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3581 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3581 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1511 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1511 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3727 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.3727 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.8152 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8152 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.4522 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.4522 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1082 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1082 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6202 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.6202 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.0629 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0629 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1255 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1255 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8172 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8172 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1082 -0.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1082 -0.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5095 -1.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5095 -1.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9732 -1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9732 -1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6945 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6945 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3949 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3949 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6319 -2.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6319 -2.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3322 -2.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3322 -2.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5530 -3.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5530 -3.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0736 -3.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0736 -3.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3733 -2.9626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3733 -2.9626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1525 -2.4890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1525 -2.4890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2882 -3.8507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2882 -3.8507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9704 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9704 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0784 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0784 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 32 30 1 0 0 0 0 | + | 32 30 1 0 0 0 0 |
| − | 27 43 1 0 0 0 0 | + | 27 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 47 2.0029 0.3736 | + | M SVB 1 47 2.0029 0.3736 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0058 | + | ID FL5FACGL0058 |
| − | KNApSAcK_ID C00005960 | + | KNApSAcK_ID C00005960 |
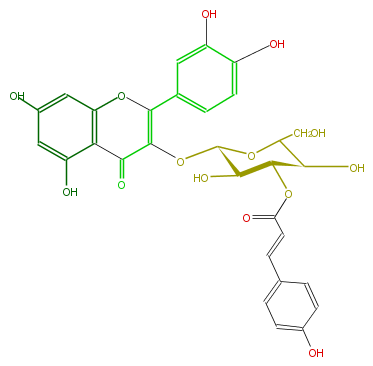
| − | NAME Quercetin 3-(3''-p-coumarylglucoside) | + | NAME Quercetin 3-(3''-p-coumarylglucoside) |
| − | CAS_RN 70286-61-2,67214-04-4 | + | CAS_RN 70286-61-2,67214-04-4 |
| − | FORMULA C30H26O14 | + | FORMULA C30H26O14 |
| − | EXACTMASS 610.13225554 | + | EXACTMASS 610.13225554 |
| − | AVERAGEMASS 610.51904 | + | AVERAGEMASS 610.51904 |
| − | SMILES c(c1)(C(=C(O[C@@H](O4)[C@H](O)[C@H](OC(C=Cc(c5)ccc(O)c5)=O)[C@@H](C4CO)O)3)Oc(c2)c(C(=O)3)c(O)cc(O)2)cc(O)c(c1)O | + | SMILES c(c1)(C(=C(O[C@@H](O4)[C@H](O)[C@H](OC(C=Cc(c5)ccc(O)c5)=O)[C@@H](C4CO)O)3)Oc(c2)c(C(=O)3)c(O)cc(O)2)cc(O)c(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.1971 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1971 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6408 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0845 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0845 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6408 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5282 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9719 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9719 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5282 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5282 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4158 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1511 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7181 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7181 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1511 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4158 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6408 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5118 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6921 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3581 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1511 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3727 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.8152 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.4522 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1082 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6202 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.0629 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1255 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8172 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1082 -0.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5095 -1.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9732 -1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6945 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3949 -1.9352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6319 -2.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3322 -2.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5530 -3.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0736 -3.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3733 -2.9626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1525 -2.4890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2882 -3.8507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9704 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0784 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 23 1 0 0 0 0
26 31 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
32 30 1 0 0 0 0
27 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 CH2OH
M SVB 1 47 2.0029 0.3736
S SKP 8
ID FL5FACGL0058
KNApSAcK_ID C00005960
NAME Quercetin 3-(3''-p-coumarylglucoside)
CAS_RN 70286-61-2,67214-04-4
FORMULA C30H26O14
EXACTMASS 610.13225554
AVERAGEMASS 610.51904
SMILES c(c1)(C(=C(O[C@@H](O4)[C@H](O)[C@H](OC(C=Cc(c5)ccc(O)c5)=O)[C@@H](C4CO)O)3)Oc(c2)c(C(=O)3)c(O)cc(O)2)cc(O)c(c1)O
M END