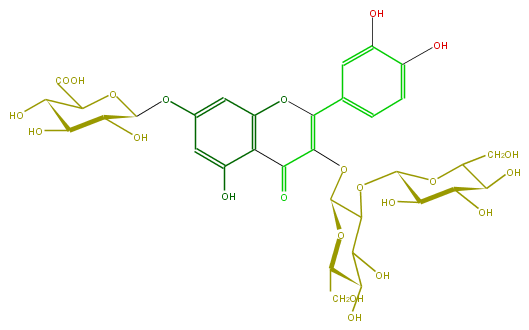
Mol:FL5FACGL0035
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5385 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5385 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5385 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5385 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8375 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8375 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1365 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1365 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1365 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1365 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8375 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8375 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5646 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5646 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2656 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2656 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2656 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2656 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5646 1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5646 1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5646 -0.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5646 -0.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9663 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9663 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6808 1.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6808 1.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3953 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3953 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3953 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3953 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6808 2.7932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6808 2.7932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9663 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9663 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2393 1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2393 1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9979 -0.0849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9979 -0.0849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8375 -0.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8375 -0.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1942 2.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1942 2.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4195 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4195 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6851 -0.8443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6851 -0.8443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9181 -1.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9181 -1.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6851 -2.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6851 -2.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4195 -2.9040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4195 -2.9040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1865 -2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1865 -2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3762 -0.5248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3762 -0.5248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8313 -2.6114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8313 -2.6114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1634 -3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1634 -3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2485 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2485 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8060 -0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8060 -0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6089 -0.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6089 -0.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3835 -0.5790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3835 -0.5790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8206 -0.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8206 -0.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1183 -0.3866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1183 -0.3866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1336 -0.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1336 -0.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2764 -1.1134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2764 -1.1134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9378 -0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9378 -0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6808 3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6808 3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0863 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0863 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5231 0.8047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5231 0.8047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7121 1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7121 1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9296 1.1286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9296 1.1286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4982 1.6973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4982 1.6973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2077 1.3228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2077 1.3228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7056 1.1905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7056 1.1905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2439 0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2439 0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9123 0.6584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9123 0.6584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8982 2.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8982 2.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9378 2.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9378 2.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3784 2.9135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3784 2.9135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6669 -3.1540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6669 -3.1540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4144 -3.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4144 -3.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4664 0.2493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4664 0.2493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2301 1.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2301 1.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 18 1 0 0 0 0 | + | 44 18 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 25 53 1 0 0 0 0 | + | 25 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 35 55 1 0 0 0 0 | + | 35 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 50 51 52 | + | M SAL 1 3 50 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 57 0.6905 -0.6905 | + | M SBV 1 57 0.6905 -0.6905 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 59 0.0183 0.6740 | + | M SBV 2 59 0.0183 0.6740 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 61 | + | M SBL 3 1 61 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 61 -0.6458 -0.2653 | + | M SBV 3 61 -0.6458 -0.2653 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0035 | + | ID FL5FACGL0035 |
| − | FORMULA C33H38O23 | + | FORMULA C33H38O23 |
| − | EXACTMASS 802.180387522 | + | EXACTMASS 802.180387522 |
| − | AVERAGEMASS 802.64102 | + | AVERAGEMASS 802.64102 |
| − | SMILES c(c6O)cc(cc6O)C(O1)=C(OC(O5)C(C(C(O)C5CO)O)OC(C4O)OC(CO)C(C4O)O)C(=O)c(c(O)2)c1cc(OC(C3O)OC(C(O)=O)C(O)C(O)3)c2 | + | SMILES c(c6O)cc(cc6O)C(O1)=C(OC(O5)C(C(C(O)C5CO)O)OC(C4O)OC(CO)C(C4O)O)C(=O)c(c(O)2)c1cc(OC(C3O)OC(C(O)=O)C(O)C(O)3)c2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-1.5385 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5385 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8375 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1365 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1365 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8375 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5646 -0.0632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2656 0.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2656 1.1511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5646 1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5646 -0.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9663 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6808 1.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3953 1.5556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3953 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6808 2.7932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9663 2.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2393 1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9979 -0.0849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8375 -0.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1942 2.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4195 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6851 -0.8443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9181 -1.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6851 -2.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4195 -2.9040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1865 -2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3762 -0.5248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8313 -2.6114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1634 -3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2485 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8060 -0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6089 -0.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3835 -0.5790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8206 -0.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1183 -0.3866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1336 -0.8701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2764 -1.1134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9378 -0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6808 3.6179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0863 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5231 0.8047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7121 1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9296 1.1286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4982 1.6973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2077 1.3228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7056 1.1905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2439 0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9123 0.6584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8982 2.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9378 2.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3784 2.9135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6669 -3.1540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4144 -3.0149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4664 0.2493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2301 1.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 18 1 0 0 0 0
50 51 2 0 0 0 0
50 52 1 0 0 0 0
46 50 1 0 0 0 0
53 54 1 0 0 0 0
25 53 1 0 0 0 0
55 56 1 0 0 0 0
35 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 50 51 52
M SBL 1 1 57
M SMT 1 ^ COOH
M SBV 1 57 0.6905 -0.6905
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 59
M SMT 2 ^ CH2OH
M SBV 2 59 0.0183 0.6740
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 61
M SMT 3 CH2OH
M SBV 3 61 -0.6458 -0.2653
S SKP 5
ID FL5FACGL0035
FORMULA C33H38O23
EXACTMASS 802.180387522
AVERAGEMASS 802.64102
SMILES c(c6O)cc(cc6O)C(O1)=C(OC(O5)C(C(C(O)C5CO)O)OC(C4O)OC(CO)C(C4O)O)C(=O)c(c(O)2)c1cc(OC(C3O)OC(C(O)=O)C(O)C(O)3)c2
M END