Mol:FL5FACGL0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9563 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9563 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9563 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9563 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4000 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4000 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8437 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8437 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8437 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8437 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4000 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4000 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2874 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7311 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7311 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7311 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7311 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 1.5643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2874 1.5643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 -0.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2874 -0.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1750 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1750 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3920 1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3920 1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9590 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9590 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9590 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9590 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3920 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3920 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1750 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1750 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5124 1.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5124 1.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3369 0.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3369 0.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4000 -0.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4000 -0.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5929 2.5849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5929 2.5849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0537 -0.9059 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.0537 -0.9059 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.5291 -0.5694 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5291 -0.5694 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3442 -1.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3442 -1.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5291 -1.8674 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5291 -1.8674 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0537 -2.2040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0537 -2.2040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1312 -1.5569 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.1312 -1.5569 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.1138 -0.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1138 -0.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3303 -1.8233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3303 -1.8233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3895 -3.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3895 -3.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0880 0.3963 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.0880 0.3963 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.5304 -0.1877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5304 -0.1877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1674 0.0601 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1674 0.0601 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7822 0.0667 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7822 0.0667 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3355 0.5135 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.3355 0.5135 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7781 0.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7781 0.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8407 -0.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8407 -0.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5598 -0.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5598 -0.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7734 -0.0654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7734 -0.0654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3920 3.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3920 3.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6389 0.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6389 0.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7989 1.6159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7989 1.6159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0475 -2.7226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0475 -2.7226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0472 -2.7012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0472 -2.7012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 43 1 0 0 0 0 | + | 25 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 47 0.0394 -2.6174 | + | M SVB 2 47 0.0394 -2.6174 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 45 2.6592 0.4897 | + | M SVB 1 45 2.6592 0.4897 |
| − | S SKP 8 | + | S SKP 8 |
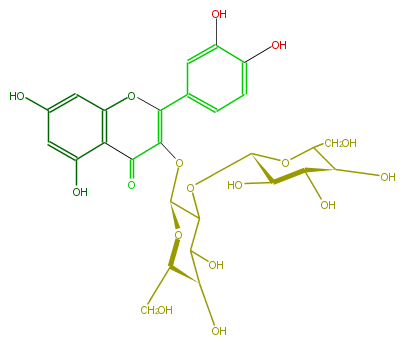
| − | ID FL5FACGL0009 | + | ID FL5FACGL0009 |
| − | KNApSAcK_ID C00005409 | + | KNApSAcK_ID C00005409 |
| − | NAME Quercetin 3-sophoroside | + | NAME Quercetin 3-sophoroside |
| − | CAS_RN 18609-17-1 | + | CAS_RN 18609-17-1 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES c(c5O)c(c(c(c5)3)C(C(=C(c(c4)ccc(O)c4O)O3)O[C@@H](C1O[C@H]([C@@H]2O)OC([C@H]([C@H]2O)O)CO)O[C@H](CO)[C@@H](C(O)1)O)=O)O | + | SMILES c(c5O)c(c(c(c5)3)C(C(=C(c(c4)ccc(O)c4O)O3)O[C@@H](C1O[C@H]([C@@H]2O)OC([C@H]([C@H]2O)O)CO)O[C@H](CO)[C@@H](C(O)1)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.9563 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9563 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4000 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8437 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8437 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4000 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 0.2796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7311 0.6008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7311 1.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 1.5643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 -0.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1750 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3920 1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9590 1.5642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9590 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3920 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1750 2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5124 1.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3369 0.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4000 -0.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5929 2.5849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0537 -0.9059 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.5291 -0.5694 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3442 -1.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5291 -1.8674 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0537 -2.2040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1312 -1.5569 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.1138 -0.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3303 -1.8233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3895 -3.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0880 0.3963 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.5304 -0.1877 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1674 0.0601 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7822 0.0667 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3355 0.5135 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7781 0.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8407 -0.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5598 -0.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7734 -0.0654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3920 3.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6389 0.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7989 1.6159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0475 -2.7226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0472 -2.7012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
35 41 1 0 0 0 0
41 42 1 0 0 0 0
25 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 CH2OH
M SVB 2 47 0.0394 -2.6174
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 CH2OH
M SVB 1 45 2.6592 0.4897
S SKP 8
ID FL5FACGL0009
KNApSAcK_ID C00005409
NAME Quercetin 3-sophoroside
CAS_RN 18609-17-1
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES c(c5O)c(c(c(c5)3)C(C(=C(c(c4)ccc(O)c4O)O3)O[C@@H](C1O[C@H]([C@@H]2O)OC([C@H]([C@H]2O)O)CO)O[C@H](CO)[C@@H](C(O)1)O)=O)O
M END