Mol:FL5FABGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9086 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9086 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9086 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9086 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2076 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2076 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4933 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4933 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4933 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4933 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2076 0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2076 0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1943 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1943 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8953 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8953 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8953 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8953 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1943 0.9857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1943 0.9857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1943 -1.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1943 -1.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5960 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5960 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3104 0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3104 0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0249 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0249 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0249 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0249 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3104 2.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3104 2.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5960 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5960 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6093 0.9855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6093 0.9855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6825 -0.6830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6825 -0.6830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2120 -1.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2120 -1.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8758 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8758 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2261 0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2261 0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2905 0.6573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2905 0.6573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3877 0.6670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3877 0.6670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0437 1.3232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0437 1.3232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8622 0.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8622 0.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7238 0.9681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7238 0.9681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1963 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1963 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4230 0.1273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4230 0.1273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4843 -1.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4843 -1.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8347 -2.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8347 -2.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8991 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8991 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9963 -1.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9963 -1.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6522 -1.1293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6522 -1.1293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4708 -1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4708 -1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3417 -1.5658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3417 -1.5658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9080 -1.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9080 -1.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1141 -2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1141 -2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5514 1.4661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5514 1.4661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5910 1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5910 1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0317 2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0317 2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8236 2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8236 2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7238 1.7518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7238 1.7518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1961 -0.8635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1961 -0.8635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3997 -0.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3997 -0.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 35 44 1 0 0 0 0 | + | 35 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 39 40 41 | + | M SAL 1 3 39 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 45 0.6892 -0.5749 | + | M SBV 1 45 0.6892 -0.5749 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 47 -0.7987 -0.4612 | + | M SBV 2 47 -0.7987 -0.4612 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 44 45 | + | M SAL 3 2 44 45 |
| − | M SBL 3 1 49 | + | M SBL 3 1 49 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 49 0.7254 -0.6977 | + | M SBV 3 49 0.7254 -0.6977 |
| − | S SKP 5 | + | S SKP 5 |
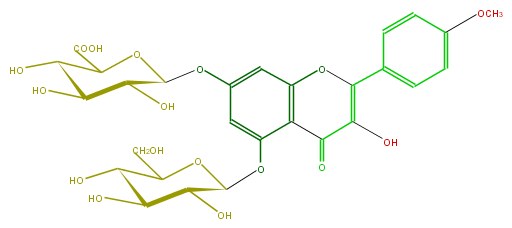
| − | ID FL5FABGS0008 | + | ID FL5FABGS0008 |
| − | FORMULA C28H30O17 | + | FORMULA C28H30O17 |
| − | EXACTMASS 638.148299534 | + | EXACTMASS 638.148299534 |
| − | AVERAGEMASS 638.5276 | + | AVERAGEMASS 638.5276 |
| − | SMILES O(C1Oc(c53)cc(cc3OC(=C(O)C(=O)5)c(c4)ccc(OC)c4)OC(C(O)2)OC(C(O)=O)C(O)C2O)C(CO)C(C(O)C(O)1)O | + | SMILES O(C1Oc(c53)cc(cc3OC(=C(O)C(=O)5)c(c4)ccc(OC)c4)OC(C(O)2)OC(C(O)=O)C(O)C2O)C(CO)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-0.9086 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9086 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2076 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4933 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4933 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2076 0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1943 -0.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8953 -0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8953 0.5810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1943 0.9857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1943 -1.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5960 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3104 0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0249 0.9855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0249 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3104 2.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5960 1.8104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6093 0.9855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6825 -0.6830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2120 -1.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8758 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2261 0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2905 0.6573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3877 0.6670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0437 1.3232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8622 0.8912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7238 0.9681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1963 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4230 0.1273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4843 -1.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8347 -2.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8991 -1.7951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9963 -1.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6522 -1.1293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4708 -1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3417 -1.5658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9080 -1.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1141 -2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5514 1.4661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5910 1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0317 2.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8236 2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7238 1.7518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1961 -0.8635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3997 -0.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 20 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
26 39 1 0 0 0 0
42 43 1 0 0 0 0
15 42 1 0 0 0 0
44 45 1 0 0 0 0
35 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 39 40 41
M SBL 1 1 45
M SMT 1 ^ COOH
M SBV 1 45 0.6892 -0.5749
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 47
M SMT 2 OCH3
M SBV 2 47 -0.7987 -0.4612
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 44 45
M SBL 3 1 49
M SMT 3 ^ CH2OH
M SBV 3 49 0.7254 -0.6977
S SKP 5
ID FL5FABGS0008
FORMULA C28H30O17
EXACTMASS 638.148299534
AVERAGEMASS 638.5276
SMILES O(C1Oc(c53)cc(cc3OC(=C(O)C(=O)5)c(c4)ccc(OC)c4)OC(C(O)2)OC(C(O)=O)C(O)C2O)C(CO)C(C(O)C(O)1)O
M END