Mol:FL5FABGI0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 59 64 0 0 0 0 0 0 0 0999 V2000 | + | 59 64 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4324 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4324 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4324 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4324 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7177 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7177 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0029 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0029 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0029 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0029 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7177 1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7177 1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7118 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7118 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4266 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4266 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4266 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4266 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7118 1.9688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7118 1.9688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7156 -0.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7156 -0.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1411 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1411 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8696 1.5480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8696 1.5480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5981 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5981 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5981 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5981 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8696 3.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8696 3.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1411 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1411 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1469 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1469 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1614 0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1614 0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7177 -0.5070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7177 -0.5070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7177 2.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7177 2.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4322 3.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4322 3.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4322 4.0314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4322 4.0314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7177 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7177 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1467 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1467 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6927 2.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6927 2.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2158 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2158 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5290 1.7140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5290 1.7140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8664 1.7212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8664 1.7212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3480 2.2028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3480 2.2028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9488 1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9488 1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2444 1.9147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2444 1.9147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9741 1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9741 1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9898 1.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9898 1.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6062 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6062 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6062 -1.5600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6062 -1.5600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0634 -1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0634 -1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7045 -0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7045 -0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7046 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7046 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2474 -0.7409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2474 -0.7409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9616 -1.9139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9616 -1.9139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0605 -1.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0605 -1.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0712 -1.3393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0712 -1.3393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9801 -1.4015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9801 -1.4015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0207 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0207 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3513 -2.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3513 -2.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1012 -2.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1012 -2.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6425 -3.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6425 -3.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3120 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3120 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5621 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5621 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5437 -1.8790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5437 -1.8790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0973 -2.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0973 -2.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5740 -2.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5740 -2.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4865 2.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4865 2.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8491 3.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8491 3.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4199 -3.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4199 -3.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9499 -4.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9499 -4.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3264 3.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3264 3.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2444 2.7003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2444 2.7003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 18 1 0 0 0 0 | + | 29 18 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 39 19 1 0 0 0 0 | + | 39 19 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 31 54 1 0 0 0 0 | + | 31 54 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 48 56 1 0 0 0 0 | + | 48 56 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 15 58 1 0 0 0 0 | + | 15 58 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 54 55 | + | M SAL 1 2 54 55 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 60 0.5378 -0.5099 | + | M SBV 1 60 0.5378 -0.5099 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 56 57 | + | M SAL 2 2 56 57 |
| − | M SBL 2 1 62 | + | M SBL 2 1 62 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 62 -0.7774 0.2397 | + | M SBV 2 62 -0.7774 0.2397 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 58 59 | + | M SAL 3 2 58 59 |
| − | M SBL 3 1 64 | + | M SBL 3 1 64 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 64 -0.7283 -0.4205 | + | M SBV 3 64 -0.7283 -0.4205 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FABGI0010 | + | ID FL5FABGI0010 |
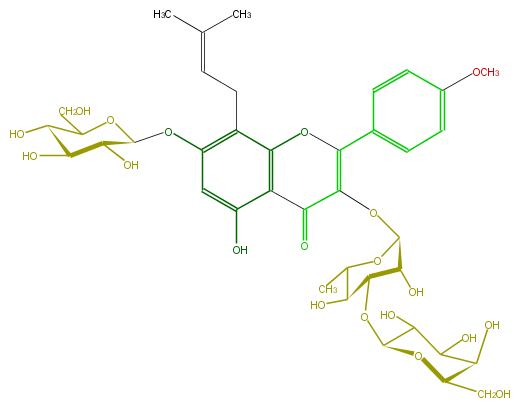
| − | FORMULA C39H50O20 | + | FORMULA C39H50O20 |
| − | EXACTMASS 838.28954404 | + | EXACTMASS 838.28954404 |
| − | AVERAGEMASS 838.8023000000001 | + | AVERAGEMASS 838.8023000000001 |
| − | SMILES O(c(c6)ccc(c6)C(O5)=C(C(c(c53)c(cc(OC(O4)C(O)C(O)C(O)C(CO)4)c3CC=C(C)C)O)=O)OC(O1)C(O)C(OC(C2O)OC(C(O)C2O)CO)C(C1C)O)C | + | SMILES O(c(c6)ccc(c6)C(O5)=C(C(c(c53)c(cc(OC(O4)C(O)C(O)C(O)C(CO)4)c3CC=C(C)C)O)=O)OC(O1)C(O)C(OC(C2O)OC(C(O)C2O)CO)C(C1C)O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
59 64 0 0 0 0 0 0 0 0999 V2000
-1.4324 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4324 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7177 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0029 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0029 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7177 1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7118 0.3180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4266 0.7307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4266 1.5561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7118 1.9688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7156 -0.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1411 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8696 1.5480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5981 1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5981 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8696 3.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1411 2.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1469 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1614 0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7177 -0.5070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7177 2.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4322 3.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4322 4.0314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7177 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1467 4.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6927 2.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2158 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5290 1.7140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8664 1.7212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3480 2.2028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9488 1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2444 1.9147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9741 1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9898 1.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6062 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6062 -1.5600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0634 -1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7045 -0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7046 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2474 -0.7409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9616 -1.9139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0605 -1.6678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0712 -1.3393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9801 -1.4015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0207 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3513 -2.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1012 -2.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6425 -3.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3120 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5621 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5437 -1.8790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0973 -2.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5740 -2.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4865 2.3956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8491 3.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4199 -3.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9499 -4.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3264 3.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2444 2.7003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 18 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
35 43 1 0 0 0 0
38 44 1 0 0 0 0
39 19 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 48 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
45 51 1 0 0 0 0
50 52 1 0 0 0 0
49 53 1 0 0 0 0
46 41 1 0 0 0 0
54 55 1 0 0 0 0
31 54 1 0 0 0 0
56 57 1 0 0 0 0
48 56 1 0 0 0 0
58 59 1 0 0 0 0
15 58 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 54 55
M SBL 1 1 60
M SMT 1 ^ CH2OH
M SBV 1 60 0.5378 -0.5099
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 56 57
M SBL 2 1 62
M SMT 2 CH2OH
M SBV 2 62 -0.7774 0.2397
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 58 59
M SBL 3 1 64
M SMT 3 OCH3
M SBV 3 64 -0.7283 -0.4205
S SKP 5
ID FL5FABGI0010
FORMULA C39H50O20
EXACTMASS 838.28954404
AVERAGEMASS 838.8023000000001
SMILES O(c(c6)ccc(c6)C(O5)=C(C(c(c53)c(cc(OC(O4)C(O)C(O)C(O)C(CO)4)c3CC=C(C)C)O)=O)OC(O1)C(O)C(OC(C2O)OC(C(O)C2O)CO)C(C1C)O)C
M END